New Phytologist—陆钰明课题组开发新型基因沉默方法
近日,上海交通大学陆钰明教授课题组在《New Phytologist》杂志在线发表了题为“In-locus gene silencing in plants using genome editing”的研究论文。该研究通过在水稻基因的非编码区引入含有上游起始密码子的调控元件(ATGE),实现了对目标基因的原位翻译沉默。经过优化,作者表明在基因起始密码子附近精准插入ATGE能够显著下调目标基因蛋白的表达。

基因表达调控技术对于作物遗传改良和粮食安全具有重要意义。目前应用最广泛的基因沉默方法包括CRISPR/Cas、RNAi、CRISPRi等技术,但由于其特性,难以实现对目标基因的可预测精细微调。大规模启动子突变虽然能将目标基因的表达调整到不同水平,但具有随机性,需要耗费大量的人力物力。因此,有必要对基因调控技术进行拓展和优化。
非编码区存在许多重要的元件,这些元件在基因表达调控和蛋白质合成中发挥关键作用。其中,上游起始密码子(upstream start codon,uATG)和上游开放阅读框(upstream open reading frame,uORF)是一类常见的表达调控元件,可以影响主要开放阅读框(primary open reading frame,pORF)的翻译。利用CRISPR/Cas技术对非编码区元件进行编辑,能够实现目标基因和敲高和敲低。
基于以上研究基础,本文作者尝试利用CRISPR/Cas技术将uATG引入到目标基因的非编码区,以期通过竞争性抑制来降低pORF的蛋白翻译。然而,uATG的引入仅在一个基因位点实现了预期的基因沉默。进一步测试发现,在目标基因的不同位置引入uATG对基因的表达产生了差异化影响,表明了其存在复杂的调控机制。因此,研究人员对原位基因沉默策略进行了一系列优化。首先,研究人员设计了多种ATGE,这些ATGE的序列源自常用的蛋白标签。原生质体瞬时实验结果表明,ATGE的引入可以有效地降低目标基因的表达水平。接下来,作者对基于HiBiT蛋白标签序列的ATGE进行了再设计,产生了带有终止密码子的ATGE1、不带终止密码子的ATGE2、带有Kozak序列的ATGE3和组合型元件ATGE4。实验结果表明,终止密码子和Kozak序列的引入能够可控地调节目标基因的表达。通过正向或反向插入ATGE4,也可实现对基因表达的可变调控。因此,作者利用靶向敲入技术将ATGE4定点插入到目标基因起始密码子上游,实现了目标基因蛋白表达从19%到89%的可调沉默,验证了抑制元件介导原位基因下调策略的可行性。通过基因编辑技术将ATGE4定点插入到Waxy基因的起始密码子上游,改变了水稻的直链淀粉含量,展示了这一技术在作物育种中的潜在价值。
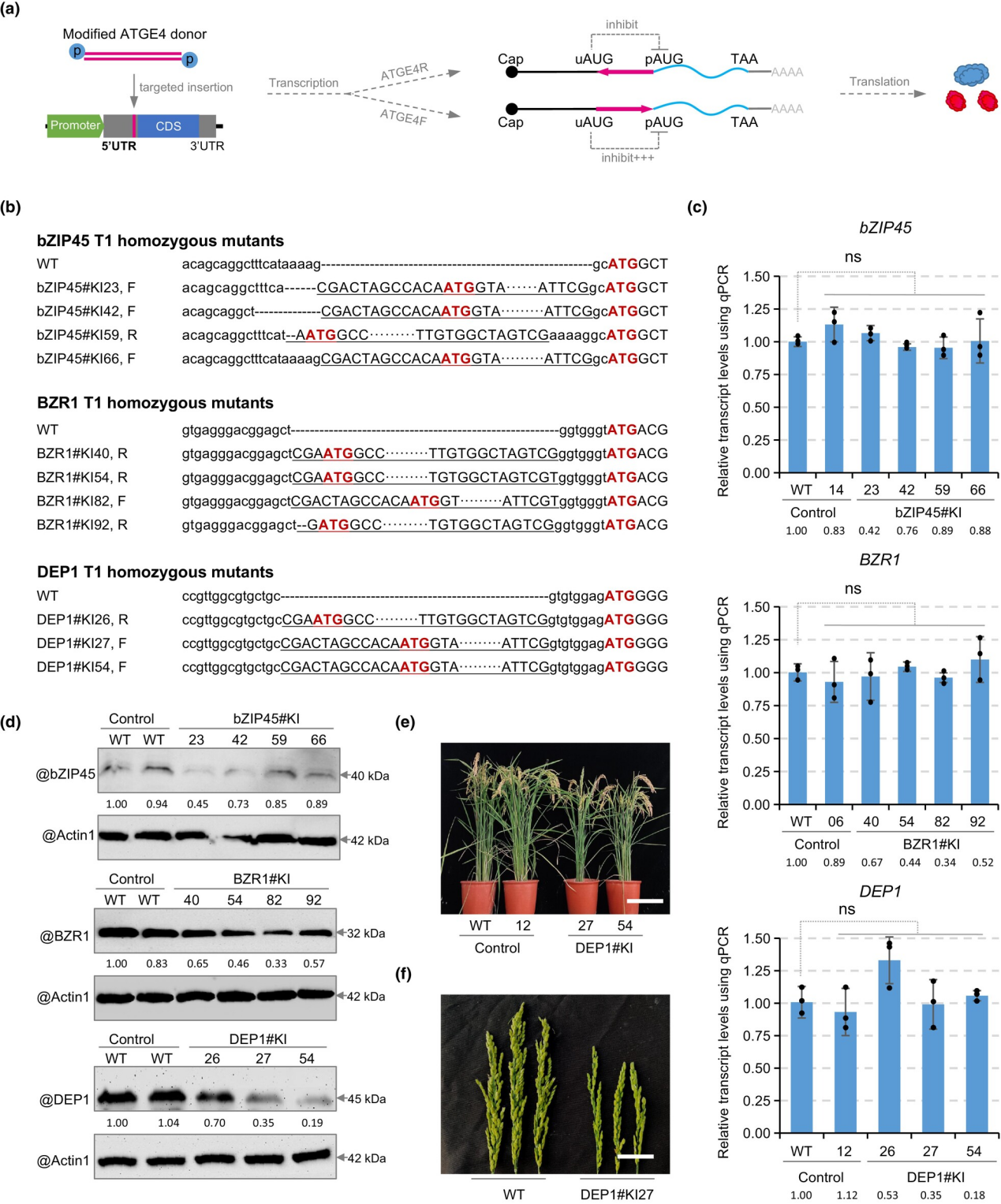
综上所述,该研究开发的原位沉默方法能够在植物特定目标基因中实现显著且稳定的表达下调,为植物科学研究和分子设计育种提供了重要的技术手段。随着精准编辑技术的不断进步,利用基因编辑技术实现mRNA翻译可控微调可能会变得更加容易。而保守的蛋白质翻译机制也为该方法在其他领域的应用提供了可能性。
沈润东博士、姚琦博士和博士研究生谭新行为论文第一作者,陆钰明教授、朱健康教授和田益夫博士为通讯作者。该研究得到了科技部重点研发计划青年项目和国家自然科学基金等项目资助。
原文连接:https://nph.onlinelibrary.wiley.com/doi/10.1111/nph.19856