服务热线
178 0020 3020
SnapGene是一款功能强大的日常分子生物学软件,旨在为用户提供完善的计划,可视化和记录您的日常分子生物学程序的解决方案。拥有友好的用户界面,可以方便,全面地记录我们经常生成的所有结构。可在SnapGene中完成所有克隆,并且优化改善你的策略,快速创建质粒图谱,并提供优雅,信息丰富的窗口,用于模拟各种常见的克隆和PCR方法。它节省了大量的时间和金钱,容易操作的可视化和模拟,提前预测可能发生的错误,并提醒用户进行改善,整个过程中,每一个操作都会自动记录,这样的话,用户可以清楚准确的看到你所有的操作内容,并可以通过.dna文件与同事共享丰富的带注释的地图和序列。
1. 界面介绍
菜单栏
File:打开、建立、保存相关文件等;
Edit:编辑功能,包括复制选中一些序列等;
View:切换几个视图等;
Enzymes:显示、选择酶切位点等;
Feature:显示、注释(命名)一些片段等;Primers:添加引物等;
Actions:进行模拟PCR、无缝克隆等;
Tools:含有序列比对等工具;
Window:窗口操作;
Help:提供帮助文件、版本号等信息。
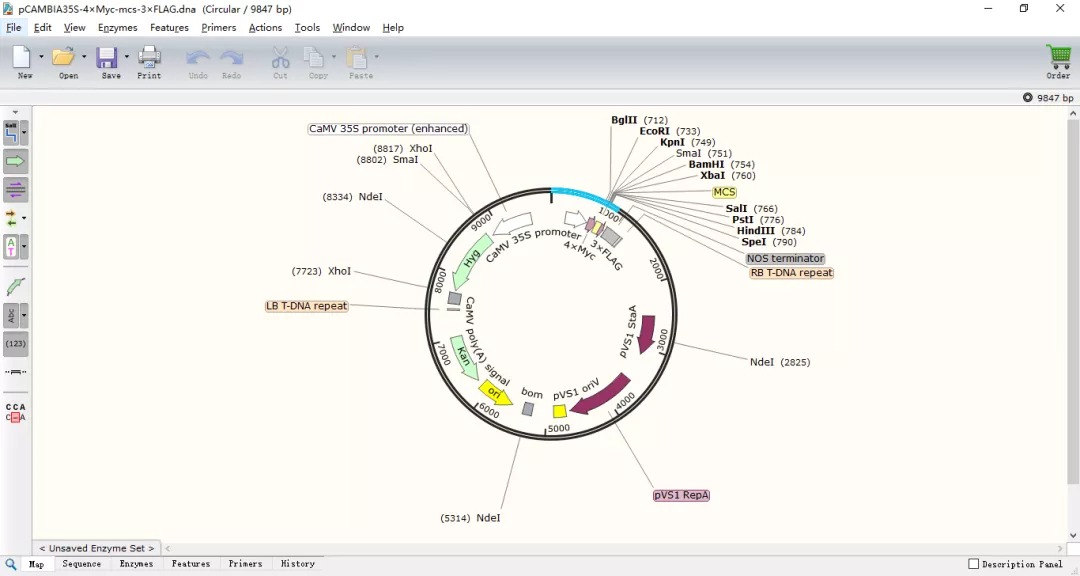
图1 pCAMBIA35S-4×Myc-MCS-3×FLAG植物表达质粒
Map视图下侧边栏
从上到下依次为:
Show enzymes:可设置限制酶的筛选条件,从而找出unique酶;
Show features:显示片段的标注;
Show primers:显示引物;
Show translations:显示翻译后的序列;
Show colors:显示自定义序列颜色(默认序列为黑色);
Show features on the DNA line:改变feature的显示位置;
Show feature labels:是否显示feature的命名;
Show locations:是否显示酶切位点的位置;
Show horiziontal map:是否将质粒以线性方式显示;
Show alignments:显示序列比对。

图2 SnapGene Map视图下侧边栏
Sequence视图下侧边栏
从上到下依次为(仅说明独特选项):
Show a minimap:在顶部显示feature;
Use compact format:仅显示文字序列;
Use 3-letter amino acid codes:氨基酸缩写转换;
Use horiziontal scrolling:单行、多行显示转换。

图3 SnapGene Sequence视图下侧边栏
2. 查看质粒
查看质粒的方式有两种:
1. dna文件可直接用SnapGne软件打开后查看
2. 网上复制的DNA序列可通过依次点击File-New DNA File或在最初界面点击DNA File后,粘贴后点击Ok即可。
3. 双元载体(binary-vector)示意图
(1)打开SnapGene,点击New DNA file,弹出图4窗口;在Creat the following sequence窗口下输入DNA序列,并对该文件命名,点击OK。或是点击Import from Genebank,输入NCBI中某序列的access number,点击OK。
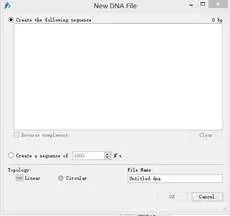
图4
(2)完成后弹出图5窗口。
(3)对该序列进行注释:点击 Features → Add Feature,对该序列进行命名注释。
图5
(4)可以根据需要点击侧边栏Show translations查看序列翻译信息。黄色箭头代表的是上面一条链编码序列,绿色箭头代表的是下面一条链编码序列。
(5)进入Sequence视图。查看Show translations下拉列表。选择All 6 frames查看所有的移码突变情况。
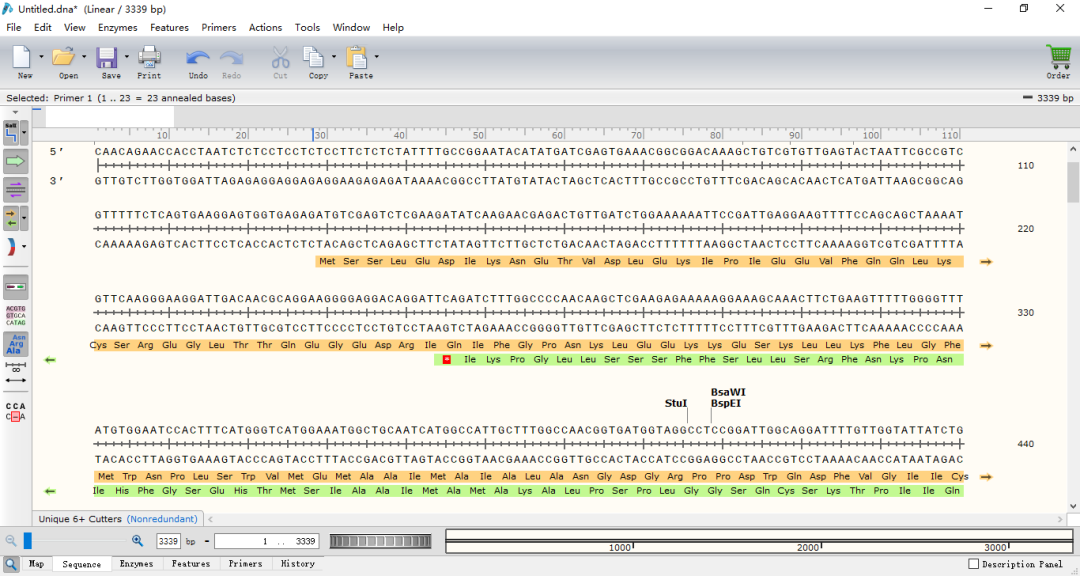
图6
4. 给目的序列添加引物及酶切位点标签
在多克隆位点处找到合适的两个酶切位点,如ApaI和XbaI。点击Primer→Add primers,将已经设计好的上游引物粘贴到5'-3'的框中。
给该引物命名,然后点击Insrtion,在该引物上添加之前选择好的酶切位点序列,如图7选择ApaI,点击Insert。GGGCCC即为酶切位点的序列。
然后在该序列5'端添加数个碱基作为保护碱基,保护碱基列表可以通过百度获取。点击OK完成添加。
下游引物设计相同,不再赘述。
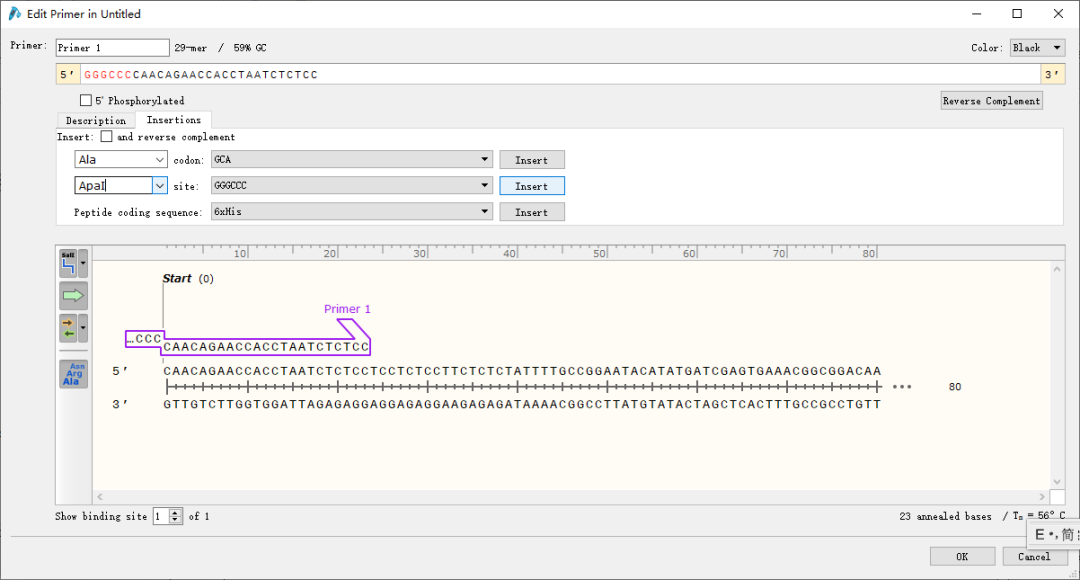
图7
5. 模拟PCR
添加引物标签的序列需要通过模拟PCR才能获得完整的Product序列。因此下面将介绍如何进行模拟PCR。
(1)点击Actions→PCR。出现图8界面,在红色框处选择引物,并命名为Amplified.dna,点击PCR。
(2)PCR完成后出现图9界面,可以看到在序列两端已经带上了我们想要的酶切位点。
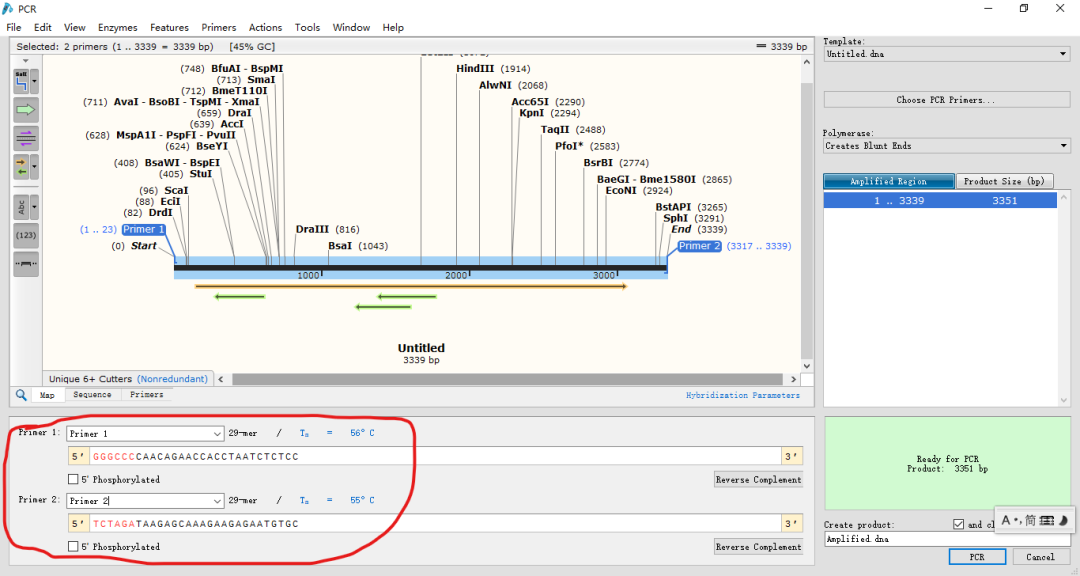
图8
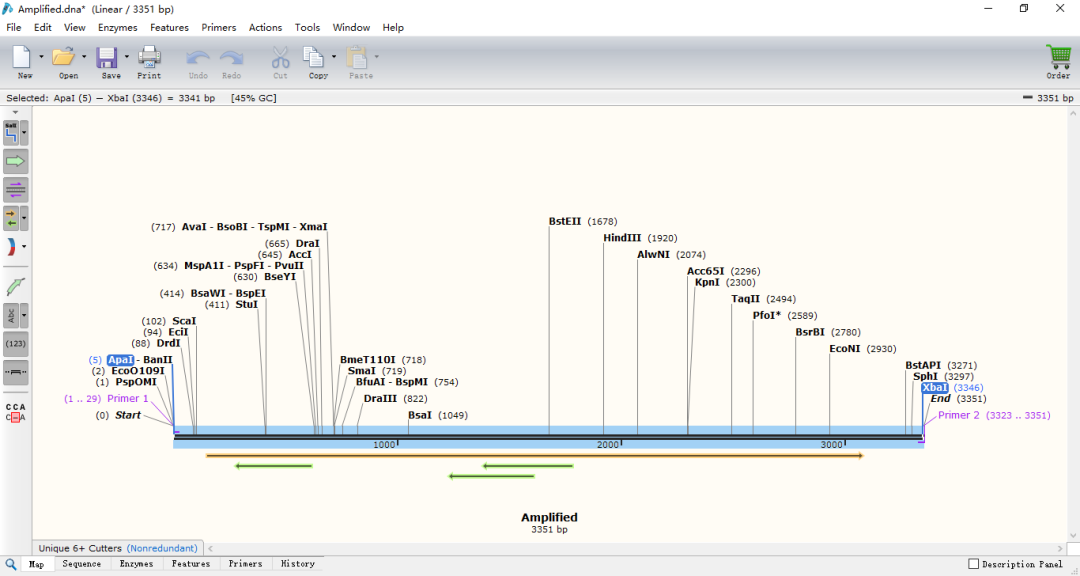
图9
6. 模拟限制性克隆
(1)打开被插入片段的质粒图谱,Ctrl+单击选择合适的两个酶切位点,如ApaI和XbaI,如图10。
(2)点击Actions→Restriction and Insertion Cloning→Insert Fragment,弹出图11窗口。
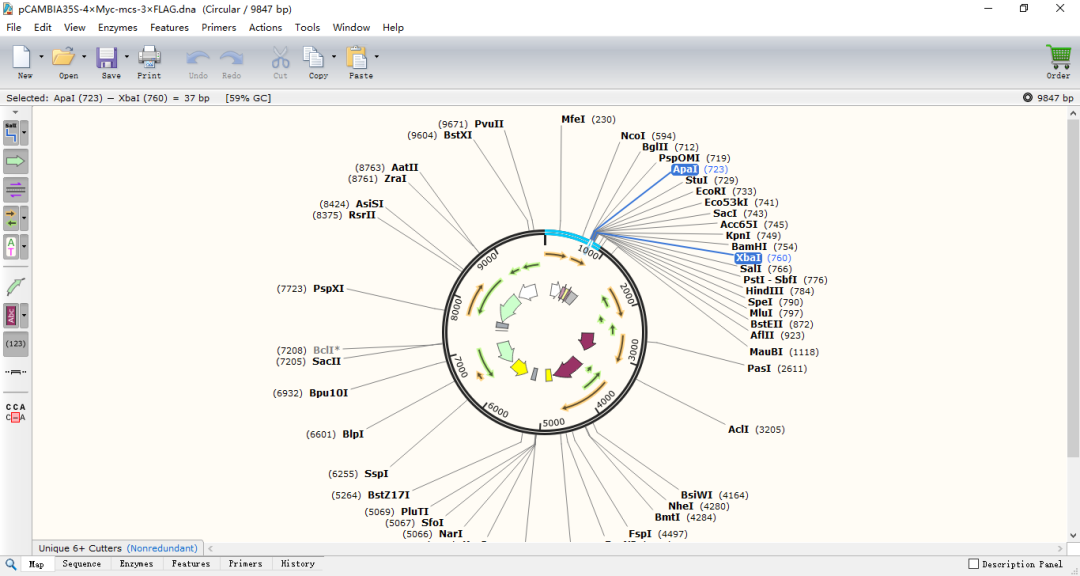
图10
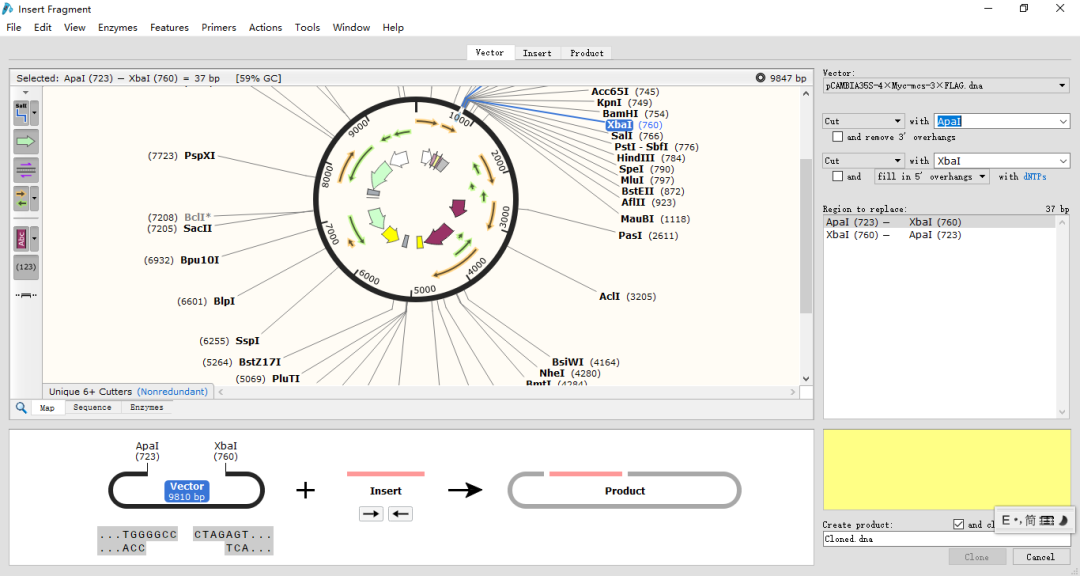
图11
(3)点击粉红色线条下面的Insert。出现图12窗口。
(4)根据顺序分别选择目的片段、限制性酶和酶切片段的长度。点击clone,出现图13窗口。保存后即完成构建。
(5)之后可根据需要标注Feature等。
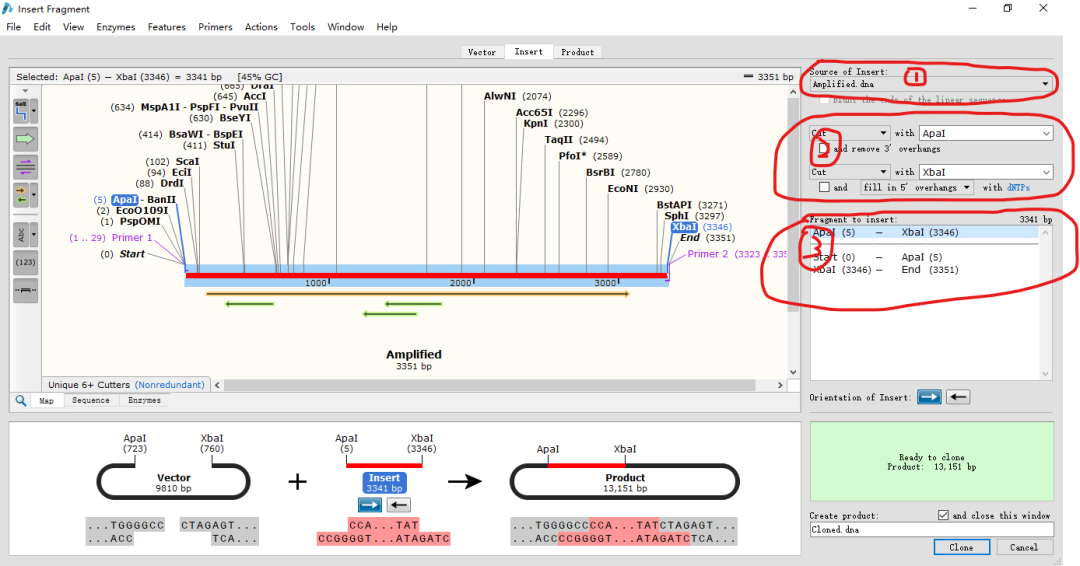
图12
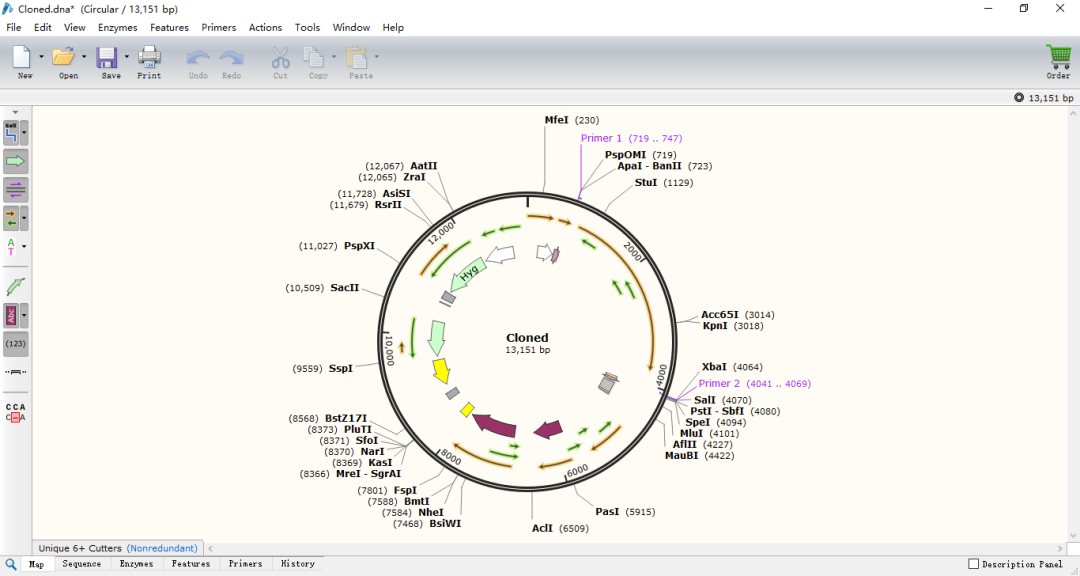
图13
附件