服务热线
178 0020 3020
以pEGFP-C1构建human p53 CDS过表达载体为例
(1)进入snapgene官网(http://www.snapgene.com/)
(2)点击主菜单栏中的“Resources”,选择“Plasmid Files”
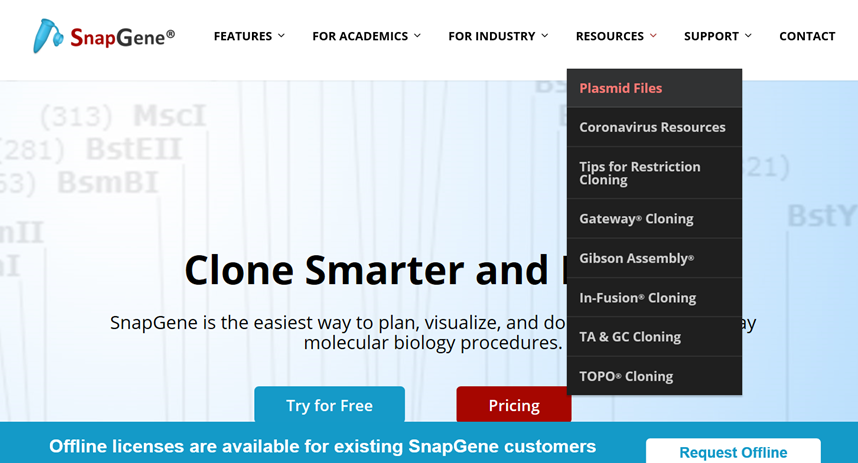
(3)在搜索栏输入载体名称“pEGFP-C1”,搜索
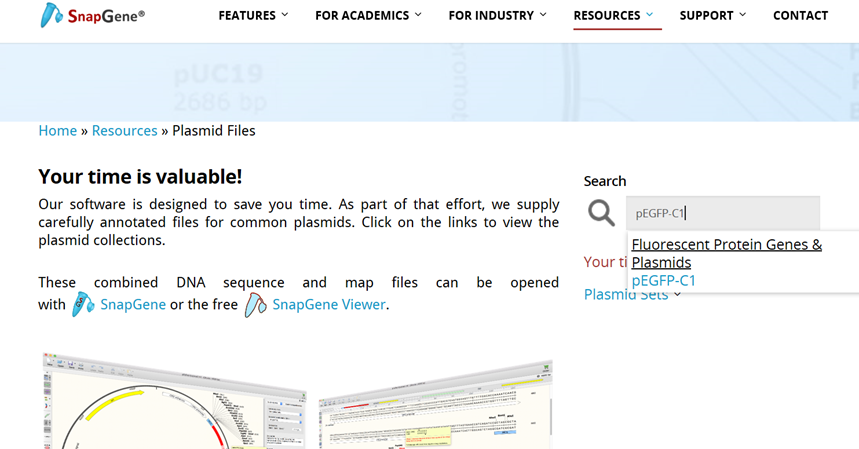
(4)如图所示,点击“Dowload”载体序列即可
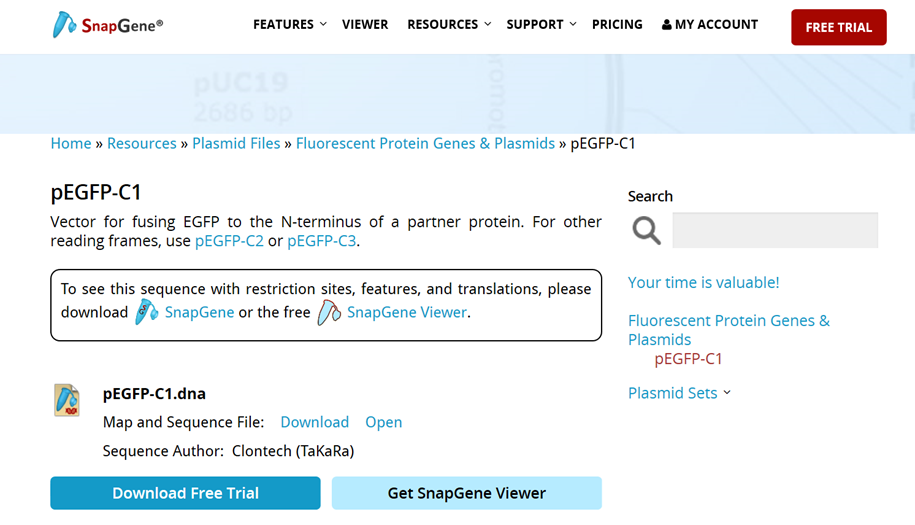
No.2
酶切位点筛选
以pEGFP-C1构建human p53 CDS过表达载体为例
(1)先从NCBI获取P53的CDS序列(NCBI官网:https://www.ncbi.nlm.nih.gov/nuccore/):进入NCBI后,选择“Nucleotide”,输入“p53”
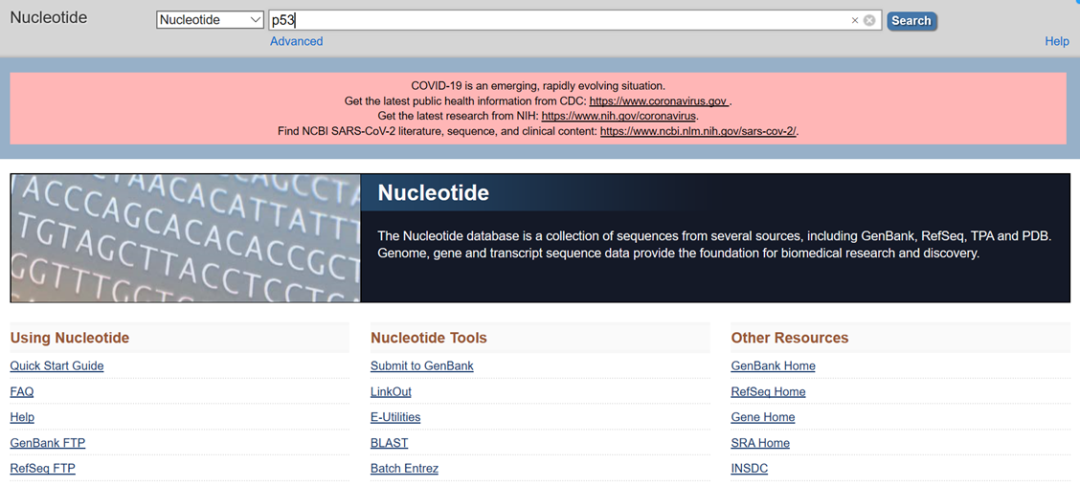
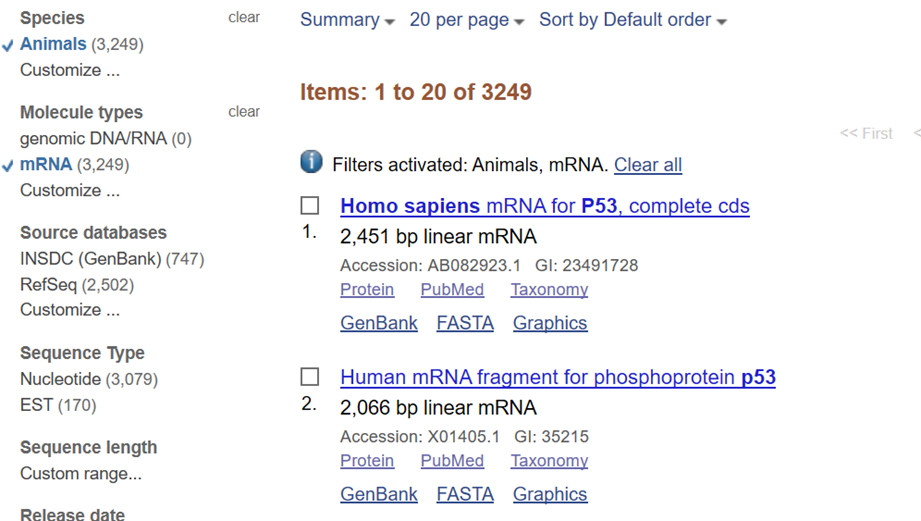
(2)在弹出的以下页面中,选择物种类别、核酸类别等进行筛选
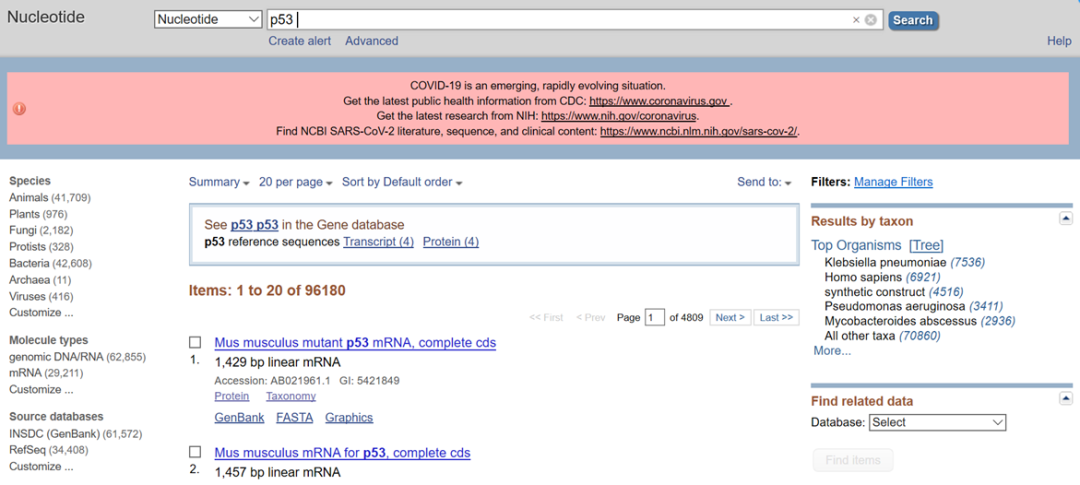
(3)点击进入置顶的这一条(也就是我们所需的人源p53 mRNA)
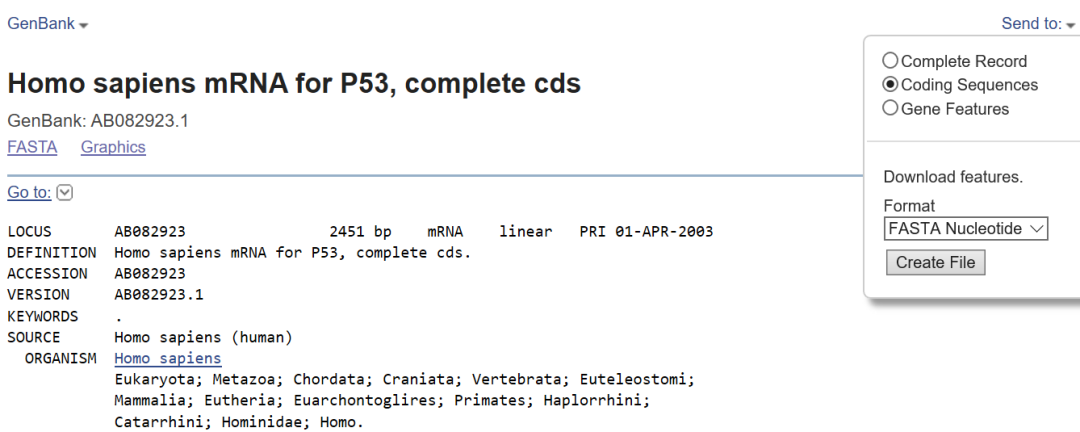
(4)点击“Send to”,选择“Coding Sequences”→“FASTA Nuleotide”,点击“Create File”,即完成目标基因(p53)CDS区序列的下载
(筛选酶切位点的方法:确定载体MCS区有哪些酶不能切割需要插入的目的基因,随后通过设计引物给目的基因两端引入这个不能切割目的基因的酶,从而完成酶切位点的筛选)
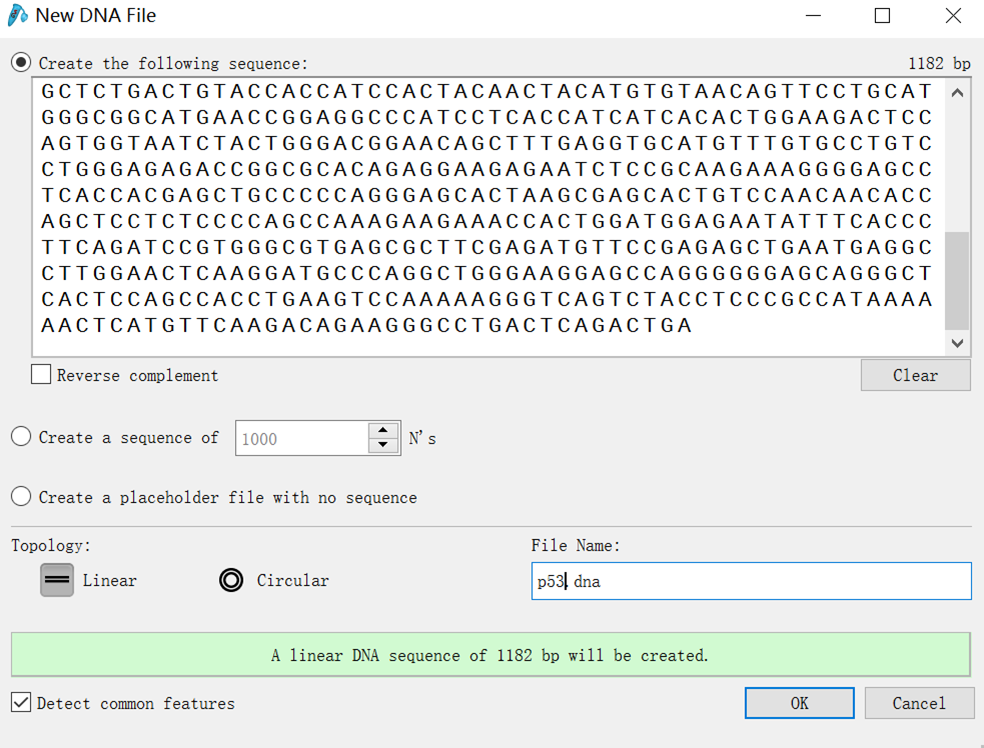
(5)打开Snapgene软件,点击“New DNA file”

(6)打开刚才我们下载的p53 CDS序列,复制粘贴到snapgene软件中,根据需要勾选相应的指标,点击“OK”
(7)我们就得到了以下界面(酶切图谱:展示了有哪些酶可以切割目标基因)
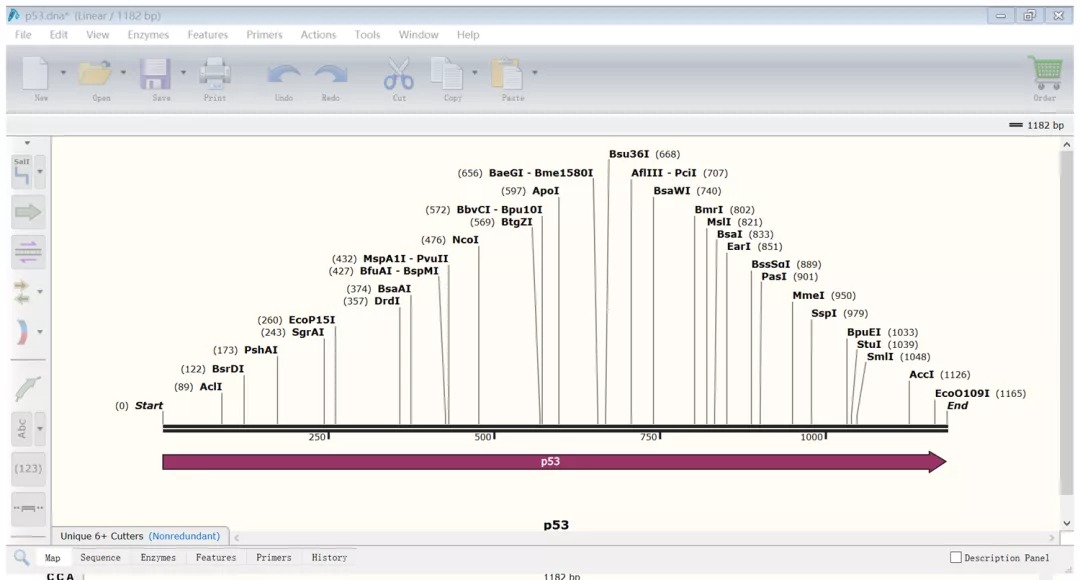
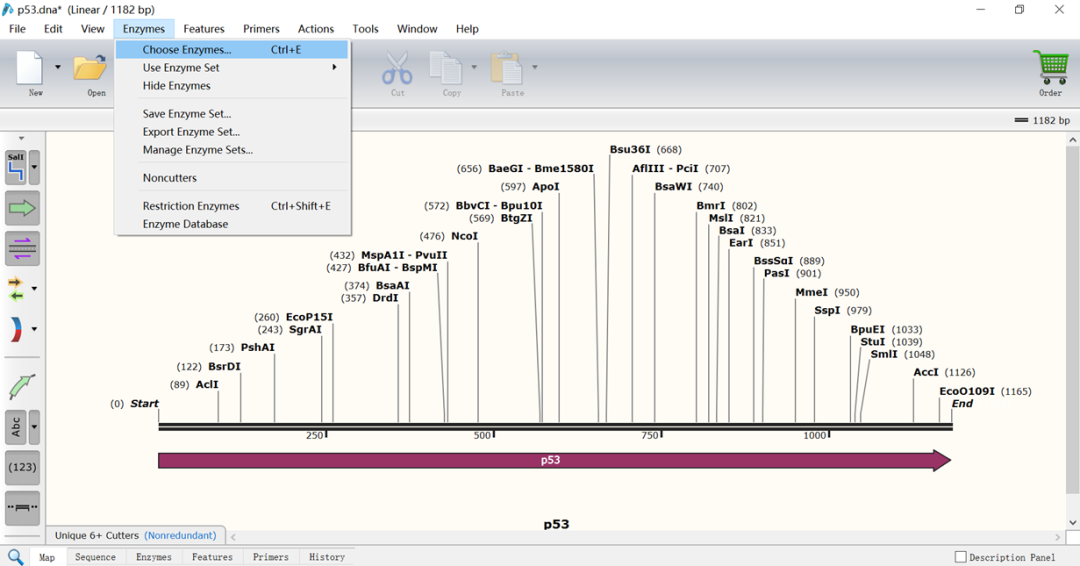
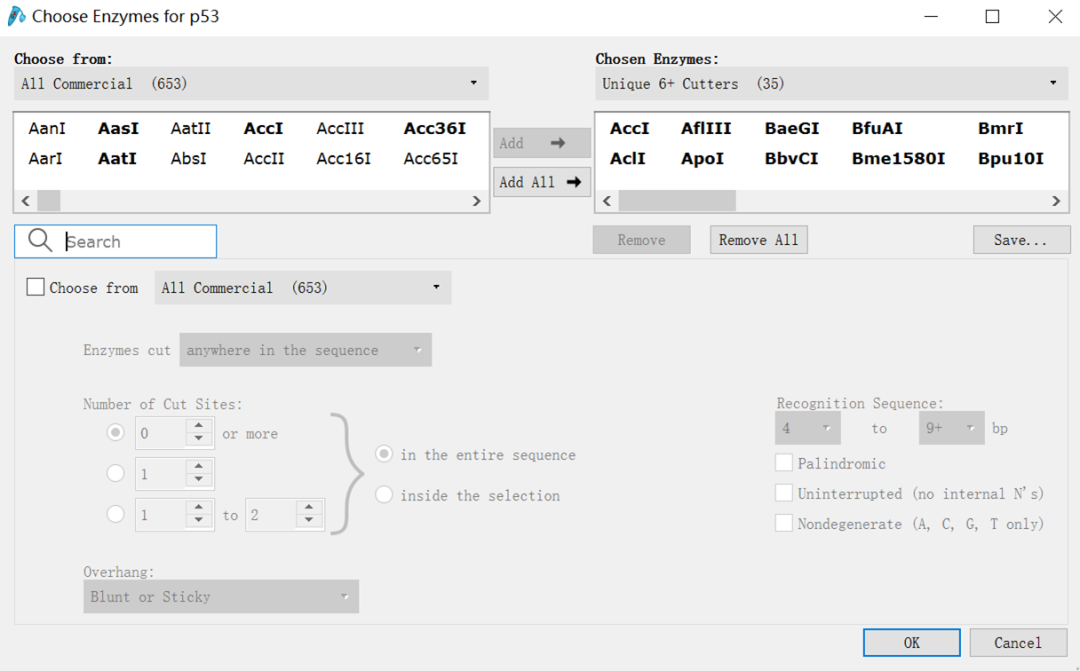
(8)将载体多克隆位点中的酶全部加入显示的图谱当中:点击“Enzymes”→“Choose Enzymes”,在弹出的新的页面中的搜索框中输入载体多克隆位点上酶的名字,点击“Add”添加到右边栏目里,点击“ok”
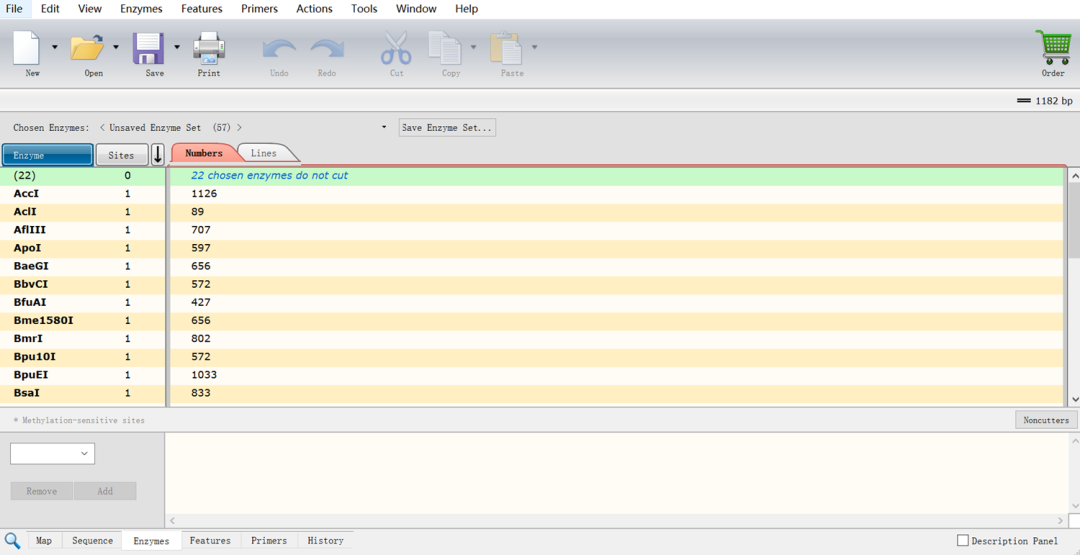
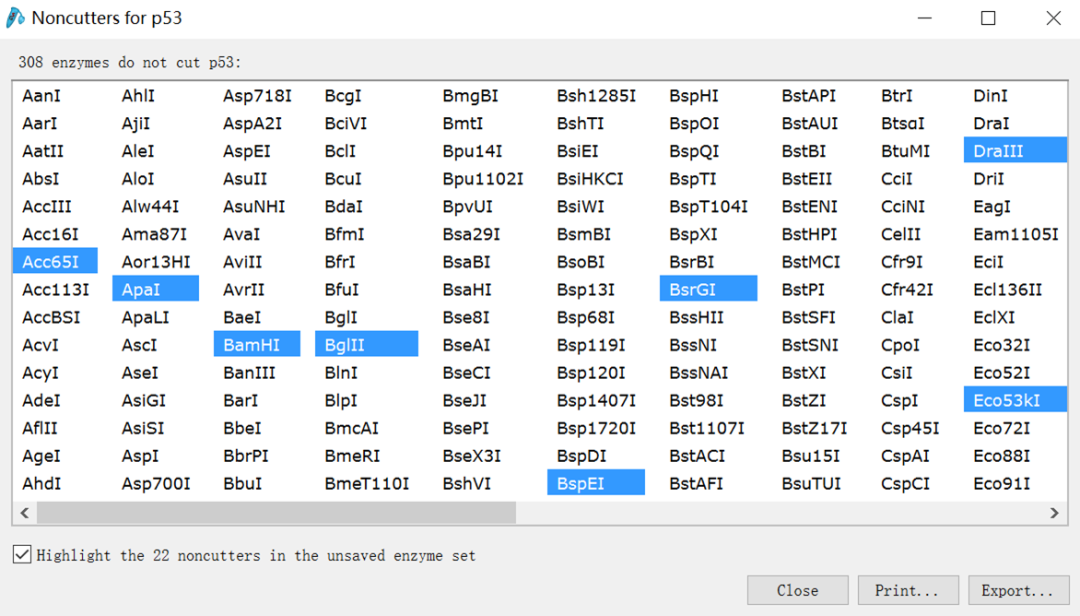
(9)点击底部菜单栏的“Enzymes”,再点击“22 chosen enzymes do not cut”,弹出的页面如下,蓝色标记的即为在载体多克隆位点出现但是在目标序列没有出现的且可以用的酶切位点
No.3
引物设计以及模拟克隆
以pEGFP-C1构建human p53 CDS过表达载体为例,通过酶切位点筛选,选择“EcoRI”和“BamHI”两种内切酶,其中需要注意的是:EcoRI在上游,BamHI在下游
(1)按照之前步骤,用snapgene打开p53 CDS序列
(2)选择底部“Sequence”页面
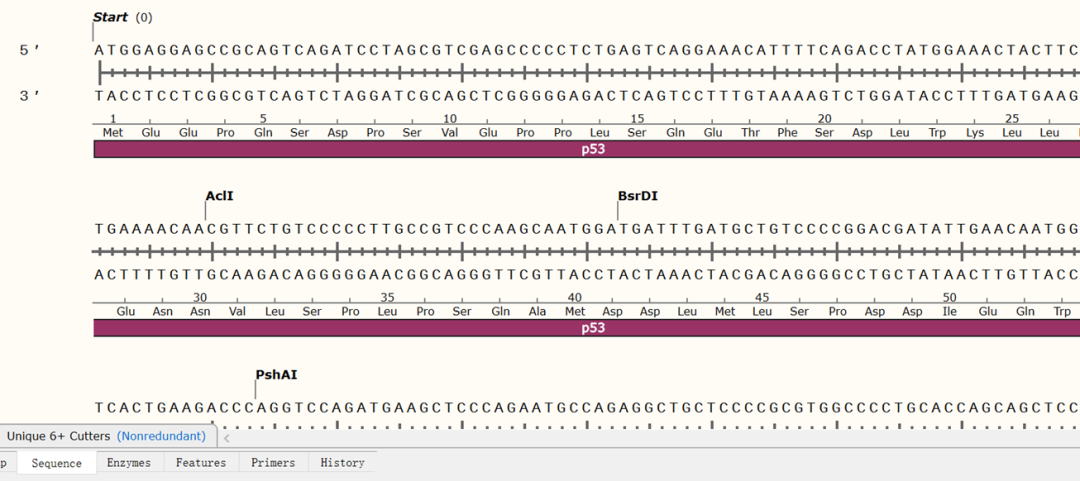
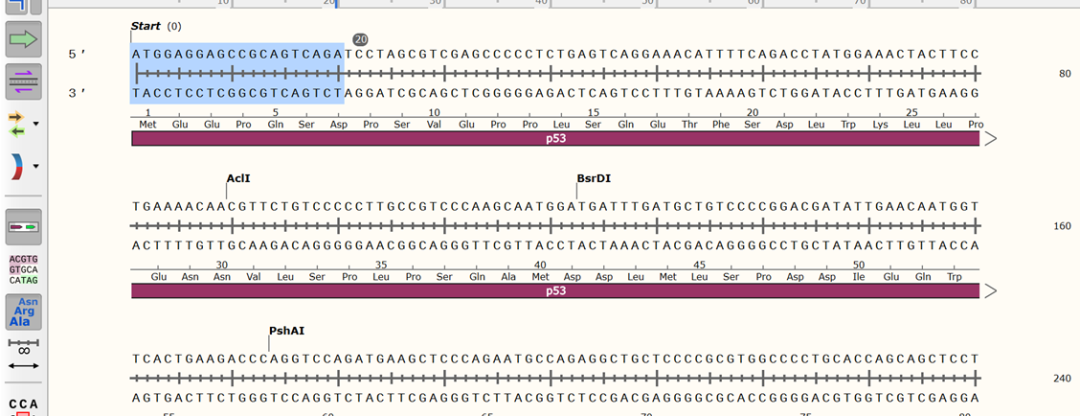
(3)接下来进行引物设计:首先先选上游前20个碱基
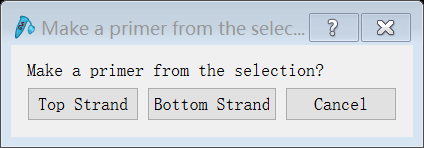
(4)点击“Primers”→“add primer”,在弹出的选项框中选择“TOP strand”
(5)按下图所示,更改上游primer的名字以及添加酶切位点序列(百度或用snapgene软件打开载体序列均可查找到内切酶对应的切割序列)和保护碱基的序列,然后点击“Add primer to template”
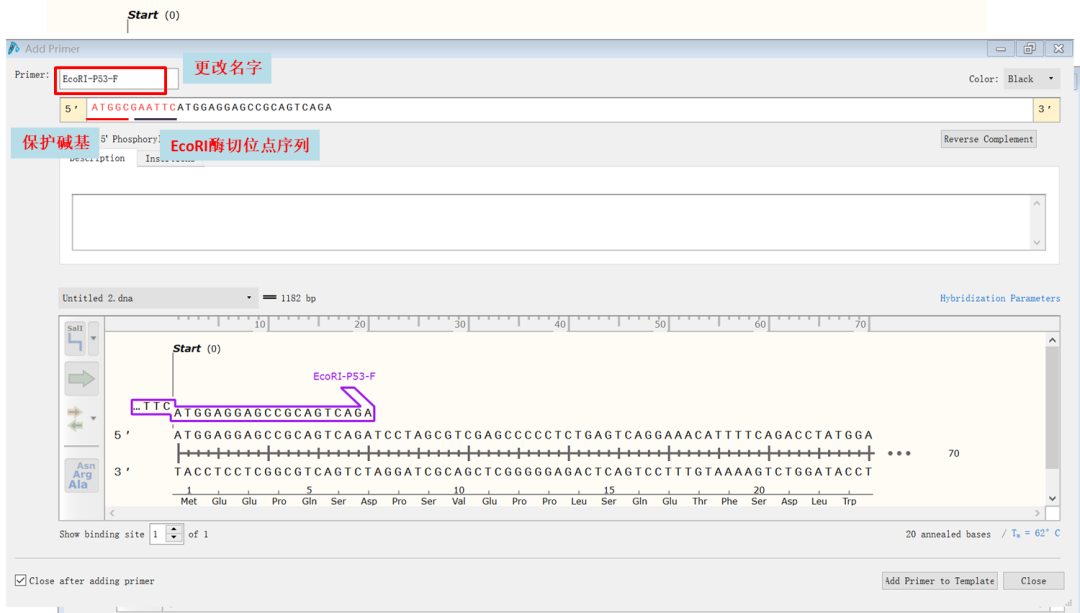
(6)然后选择下游20个碱基,点击“Edit”→“copy bottom strand”,选择“5’→3’”
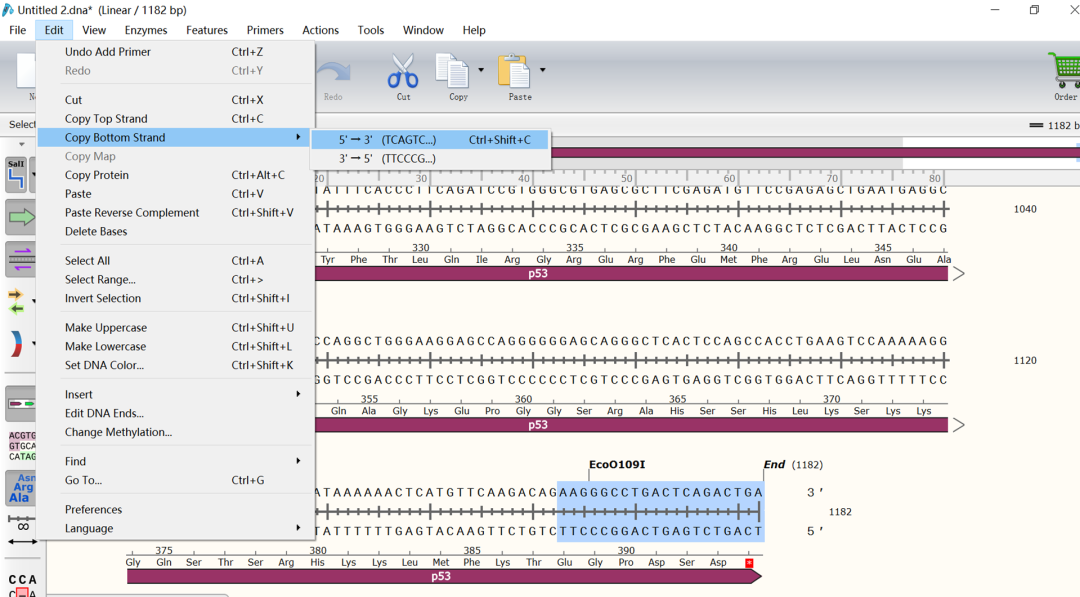
(7)然后,点击“Primers”→“Add primer”,在弹出的选项框中点击右上角的×,直接关闭
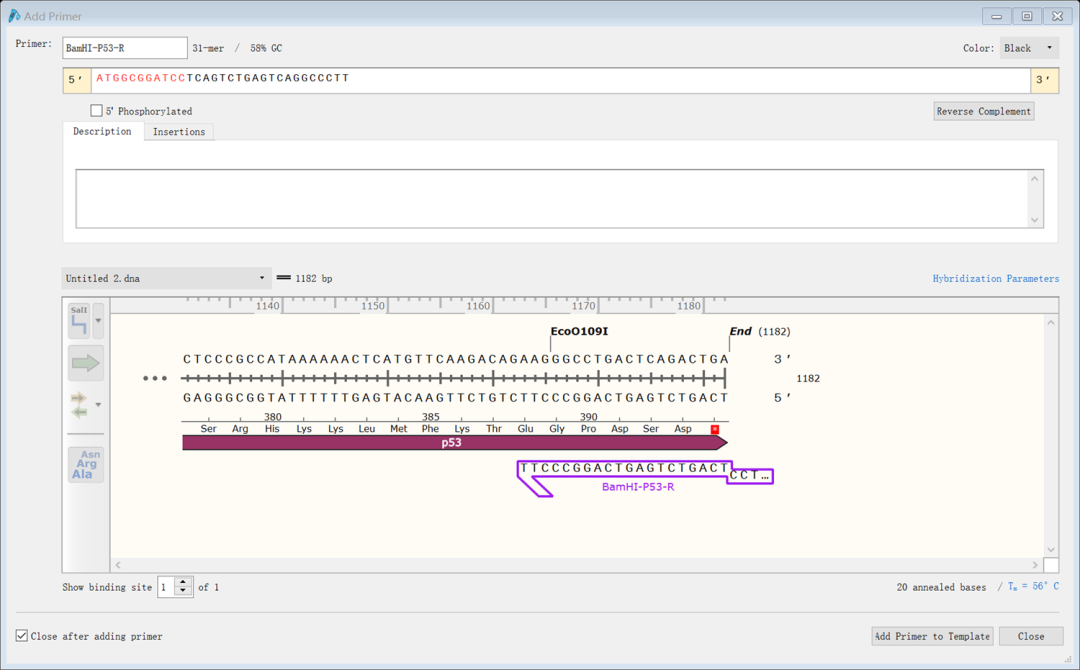
(8)将序列粘贴到序列框中,同理更改名字以及添加酶切位点序列(下游酶切位点是BamHI)和保护碱基的序列,然后点击“add primer to template”
(9)点击“Map”,Ctrl键选择两个引物(如图),点击“Action”→“PCR”
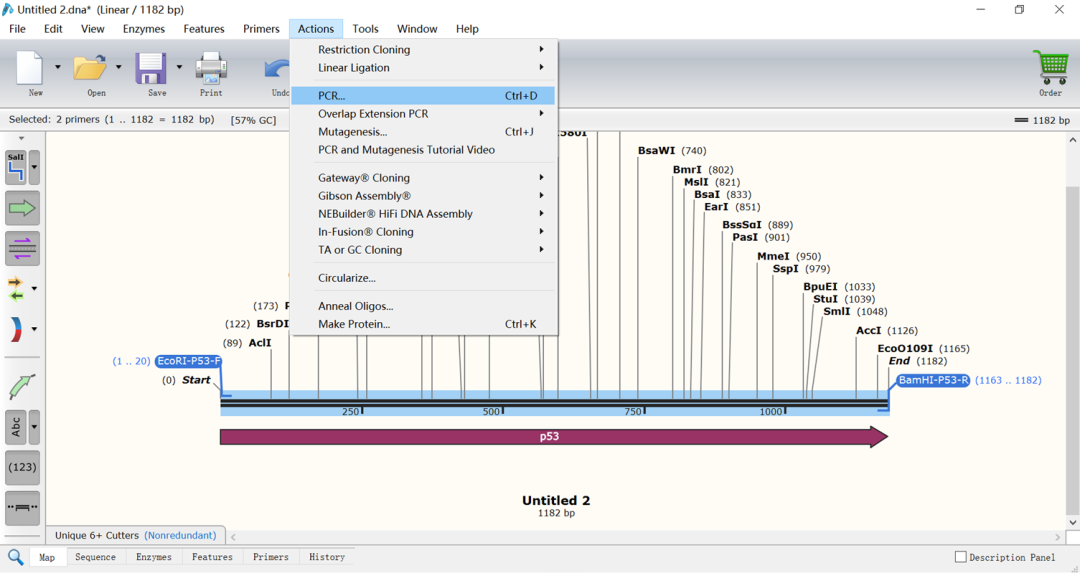
(10)点击“PCR”,即获得扩增产物(如下图所示)
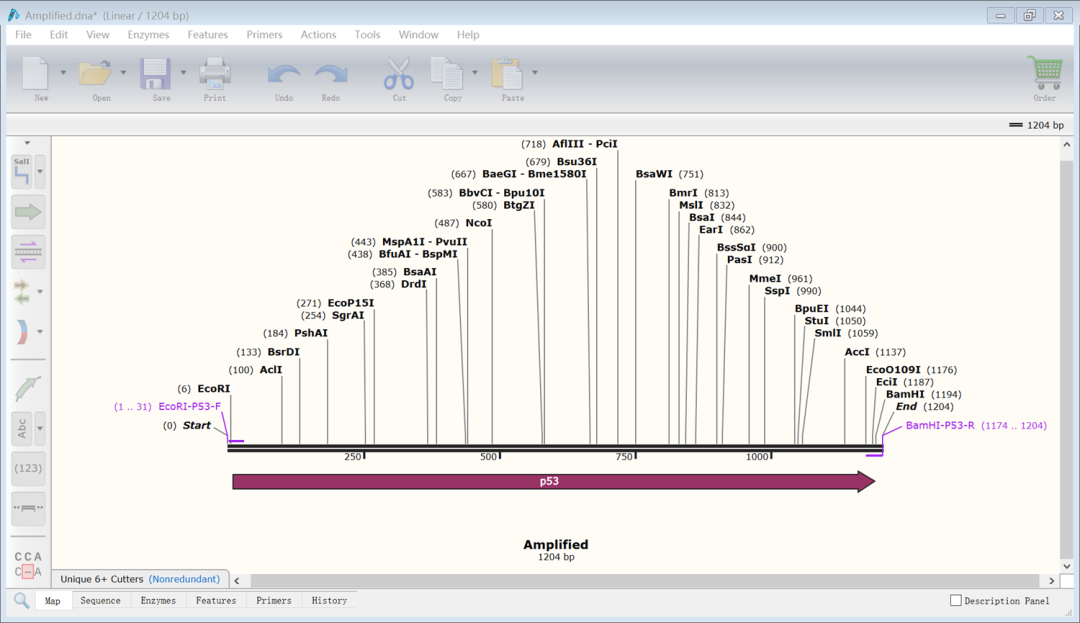
(11)打开表达载体序列,按Ctrl键选择两个酶切位点(蓝色标记的),点击“Action”→“Restriction cloning”→“Insert fragment”
(12)点击“Insert”,选择刚刚扩增产物的文件“Amplified.dna”,然后分别输入相应内切酶的名字,点击插入的片段
(13)最后在右下角点击“Clone”即可,结果如下
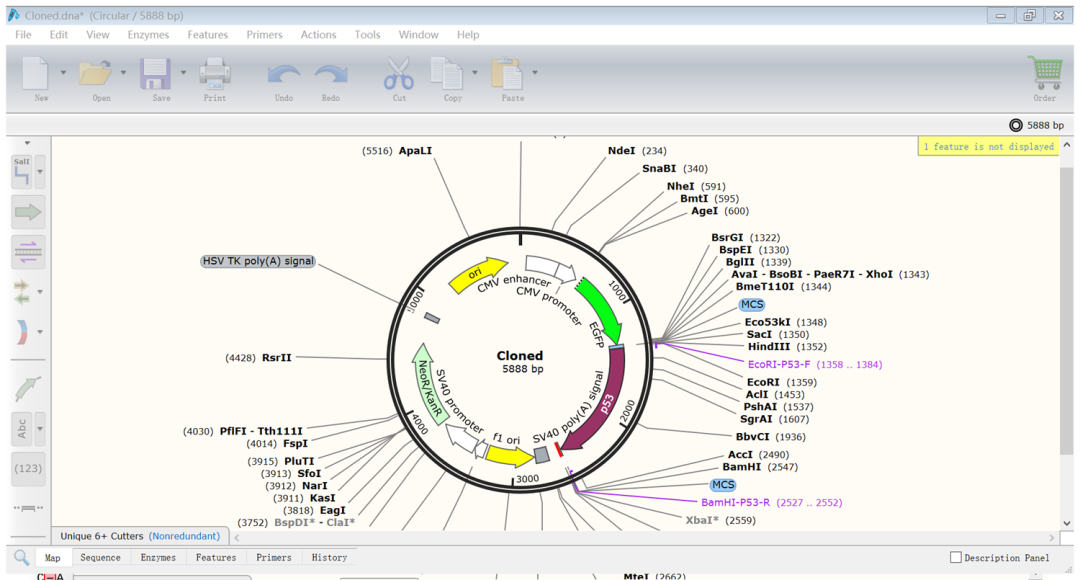
附件