NAR | 季雄团队揭示RNA聚合酶II降解导致GPCR配体结合基因上调机制
DNA损伤常导致RNA聚合酶II (Pol II) 蛋白的降解,化学物质抑制转录、UV照射,以及血清饥饿也可引发该蛋白的降解。然而,Pol II蛋白水平下降后,其具体的分子调控网络变化和相关机制仍不清楚。季雄团队早前利用蛋白质瞬时降解系统,研究了Pol II的全部12个亚基的具体功能分工。结果发现大多数亚基的降解会导致全局的基因表达下调,但意外地观察到一小部分基因表达上调的情况。本研究在此基础上进一步探讨了Pol II大亚基蛋白的降解导致转录水平上调基因的特征,阐明了这些基因表达上调的原因。此外,本研究还关注外界环境刺激条件下引起Pol II降解的情形,揭示类似的调控机制也可能会发生。

2024年6月6日 ,北京大学生命科学学院、北大-清华生命科学联合中心季雄课题组在Nucleic Acids Research发表了题为“Increased transcriptional elongation and RNA stability of GPCR ligand binding genes unveiled via RNA polymerase II degradation”的研究论文。本研究的结果显示Pol II的大亚基RPB1蛋白降解会导致全局性的基因表达下调,但与G蛋白偶联受体(GPCR)配体结合相关的一小部分基因却会上调表达,原因是这些基因的转录延伸和mRNA稳定性增加。在外部环境刺激引起Pol II的大亚基降解后,这一现象同样被观察到,表明细胞通过提高GPCR配体结合相关基因的转录延伸和RNA稳定性来应对细胞中转录全局下调引发的反馈机制。
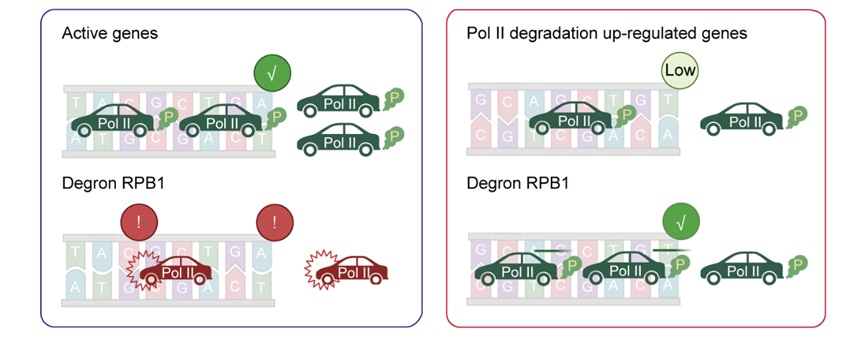
图1. 瞬时降解 Pol II 的 RPB1,尽管活跃基因的表达全局下调,部分参与GPCR配体结合相关基因反而表达增加。
外界环境刺激条件下,通常会造成Pol II 大亚基的蛋白不稳定,为了深入研究相关基因调控机制。本研究首先利用生长素诱导的蛋白瞬时降解系统对Pol II的大亚基RPB1进行降解。结果表明,RPB1降解导致了预期的全局转录抑制以及 RNA 代谢因子和染色质调控因子在染色质上的结合下降。令人惊讶的是, 少部分基因的表达呈现上调。多种转录组实验方法的结果证实,这些基因在成熟RNA和新生RNA水平均表现出明显的表达上调。这一现象在RPB2和RPB6降解后没有观测到,但是在除了RPB4和RPB12外的亚基降解后都观测到了类似现象。
Pol II 降解后上调基因特征分析发现Polycomb 结合的、较少转录暂停的短基因表达增加。这些上调的基因在参与 GPCR 配体结合的功能类别中富集。接下来,研究发现,在RPB1降解后,尽管上调基因上总的Pol II占位下降,但pSer2形式的RPB1(转录延伸标志)的占位却增加了。同时,上调基因的RNA稳定性也显著增加。这表明细胞通过提高这些基因的转录延伸和RNA稳定性,来应对RPB1降解所带来的应激反应。之后,研究团队在不同的应激条件下,如α-鹅膏蕈碱或紫外线处理,同样发现RPB1降解导致全局基因转录抑制,同时也激活了一些特定的基因。通过对这些基因的功能富集分析,发现它们同样主要参与GPCR配体结合和环境刺激响应,因而这些发现对于理解细胞在应对外部压力时的基因调控机制具有重要意义。
本研究还发现,RPB1降解后,H3K27me3占位减少而H3K27me3的“eraser”,KDM6B占位增加。RPB2降解却导致H3K9me3和H3K9me3的“writer”,SUV39H1占位减少。这表明RPB1和RPB2在染色质修饰和基因激活方面具有不同的作用机制。但是这些表观修饰变化,以及转录延伸速度、转录终止、RNA剪接和RNA 3’加工和RNA出核转运等,都不能解释基因表达的上调现象。
总体而言,本研究揭示了在RPB1蛋白降解后,特定基因通过增加转录延伸和RNA稳定性来维持其表达,这为理解细胞在面对转录抑制时的应对机制提供了新视角。特别是,GPCR配体结合基因的上调可能代表了一种细胞适应转录机器受损的应激反应机制。此外,本研究中Pol II瞬时降解后的染色质蛋白质组变化结果也为未来研究Pol II偶联的RNA、DNA代谢过程,提供了重要线索。

图2. RPB1在α-鹅膏蕈碱或紫外线处理后降解,导致全局转录抑制和特定基因上调,特定基因通过增加转录延伸和RNA稳定性来维持其表达来应对RPB1降解所带来的应激反应。
北京大学生命科学学院、北大-清华生命科学联合中心季雄研究员和北京大学生命科学学院,北京大学核糖核酸北京研究中心 (BEACON) 黄捷博士是该论文的共同通讯作者。北京大学生命科学学院博士研究生包丽君(2021级)、朱峻毅(2021级)是该论文的共同第一作者。该工作得到国家自然科学基金原创探索项目、北大-清华生命科学联合中心、启东创新基金、成都研究院前沿创新基金、细胞增殖与分化教育部重点实验室和科技部国家重点研发计划的资助。感谢北京大学凤凰工程多个仪器平台对本项目的大力支持。
参考文献:
1. Jiang, Y., Huang, J., Lun, K., Li, B., Zheng, H., Li, Y., Zhou, R., Duan, W., Wang, C., Feng, Y. et al. (2020) Genome-wide analyses of chromatin interactions after the loss of Pol I, Pol II, and Pol III. Genome biology, 21, 158.
2. Li, Y., Huang, J., Zhu, J., Bao, L., Wang, H., Jiang, Y., Tian, K., Wang, R., Zheng, H., Duan, W. et al. (2022) Targeted protein degradation reveals RNA Pol II heterogeneity and functional diversity. Molecular cell, 82, 3943-3959.e3911.
3. Huang, J. and Ji, X. (2023) Never a dull enzyme, RNA polymerase II. Transcription, 14, 49-67.
季雄课题组长期从事RNA聚合酶非经典功能调控研究。主要集中在RNA聚合酶亚基未知功能调控、分子探针和生物计算等方向,近5年成果发表在Cell(2023)、Molecular Cell(2022,2023)、Genome Biology(2020,2022)、Nature Communications (2022)、Nucleic Acids Research (2023,2024)、Cell Reports (2023)、Cell Discovery (2020)、CMLS(2022)、iScience(2022)、Transcription (2023)、STAR Protocol(2023a, 2023b)等杂志上,为选择性基因表达调控提供新的假说。欢迎感兴趣的博士后和研究生联系并申请加入。