Nucleic Acids Research|同济大学王晨飞课题组合作建立肿瘤微环境单细胞转录组学数据库TISCH2
肿瘤的发生发展过程中,其所处的免疫微环境具有高度异质性,并和疾病的发展密切相关。单细胞转录组测序技术的出现,极大地提高了肿瘤微环境研究的分辨率,使得研究人员可以在单细胞层面探究肿瘤微环境中的细胞组成以及基因表达调控的异质性。肿瘤免疫疗法在控制肿瘤方面疗效显著,但临床试验表明,由于高度复杂的肿瘤微环境,免疫疗法仅对部分患者有效。因而,利用单细胞测序技术解析不同治疗条件和治疗响应情况下肿瘤样本的微环境和生物学特征,将有助于确定导致耐药的细胞和提高肿瘤免疫疗法的实用性[1]。2020年,王晨飞课题组与合作者团队收集并整理了肿瘤病人组织的单细胞转录组数据,建立了大规模肿瘤免疫单细胞数据库TISCH[2]。TISCH发表后不到两年的时间内,已经拥有超过2万6千个用户,被引用频次超过180余次。
近日,同济大学王晨飞课题组和四川大学华西口腔医院李太文课题组再次合作在Nucleic Acids Research杂志上发表文章TISCH2: expanded datasets and new tools for single-cell transcriptome analyses of the tumor microenvironment,更新了TISCH数据库,涵盖了3倍于TISCH1的数据,并且提供了更为丰富的分析功能。

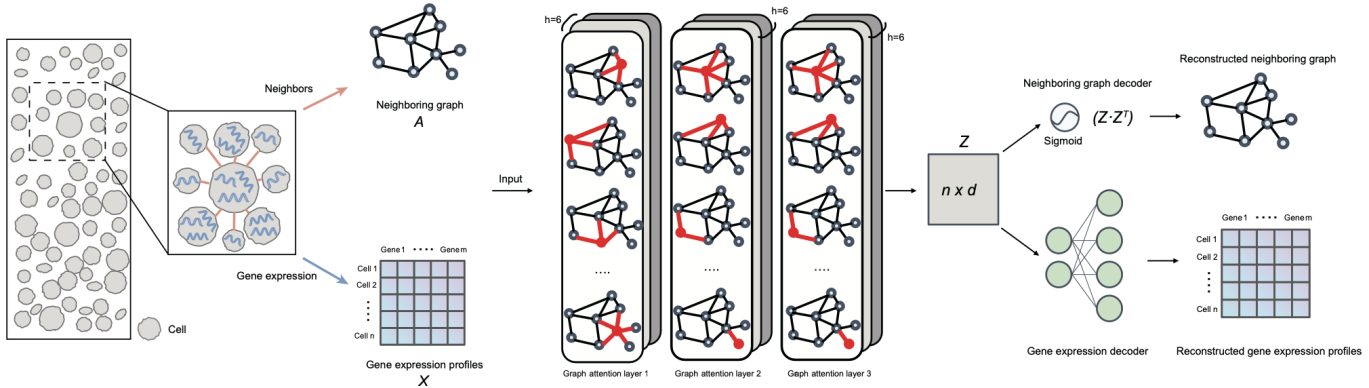
目前为止,TISCH2收集了来自GEO和ArrayExpress的共187套高质量肿瘤单细胞转录组数据以及相应的病人信息,数据涵盖了50种癌症类型,收录了来源于超过1500个病人6百万个细胞。其中TISCH2有40套数据是在不同治疗条件下的单细胞转录组数据,其中包括免疫治疗、化疗、靶向治疗和联合治疗。所有收集的数据都使用MAESTRO软件[3]进行了统一的处理分析,处理步骤包括质量控制、批次效应校正、聚类、差异表达分析、癌细胞划分、细胞类型注释、功能富集分析等(图1)。标准化的处理流程使得不同来源的数据相对可比,有利于数据的再利用。TISCH2提供了用户友好的访问界面,用户可以对肿瘤微环境进行交互式的探索。

图 1 TISCH2数据库中的数据处理流程以及功能特征概览,红色部分为新增功数据及功能
TISCH2提供了两个功能模块,数据集模块和基因模块。通过数据集模块,用户可以搜索感兴趣的数据集并进行深入的探究。为方便用户再利用数据,TISCH2对每套数据都提供了单细胞水平的表达谱以及相应的细胞类型注释、样本信息以供下载。同时,TISCH2支持多套数据互相比较,以探究不同数据集间的细胞组成以及基因表达的差异性。对于每套数据集,TISCH2提供了丰富的可视化结果以反映肿瘤微环境的细胞组成,用户可以研究基因在不同细胞、不同治疗组的表达分布特征、细胞的功能状态等信息。对于免疫治疗的数据,TISHC2提供了不同的治疗或者响应组之间细胞组成的比较、差异表达基因的分析、免疫治疗相关基因集合的表达。除了基础的功能外,TISCH2 还提供了细胞之间相互作用和转录调控的分析结果,文章以一套头颈癌[4]的数据为例,展示了数据集模块的具体用法。为了识别细胞类型特异的转录因子,TISCH2使用LISA算法[5]来推测在每种细胞群中调控基因表达的转录因子,并使用热图来展示数据集中所有细胞群各自的关键转录因子(图2A)和每个细胞群内部的关键转录因子(图2B)。肿瘤微环境中细胞间的相互作用,不仅可以调控细胞自身的功能,也会影响周围的免疫微环境从而影响肿瘤的发生发展。TISCH2使用CellChat[6]来推测不同细胞间的相互作用情况,和展示了细胞群之间两两相互作用的基因对的数目(图2C),同时描绘了特定细胞群和其他细胞群的相互作用的概率(图2D)和具体的基因对之间相互作用的概率(图2E)。

图 2 TISCH数据集模块功能展示
在TISCH2的基因模块中,研究人员可以输入一个感兴趣的基因并选择癌症类型,TISCH2就可以迅速返回不同的可视化结果用以展示该基因整体的表达情况、在TCGA 各种癌症类型中的风险比(图2F)、以及与该基因共表达其他潜在基因(图2G)。这些功能使得研究人员可以快速定位某一基因在不同细胞类型、不同癌症类型中的表达情况,以及与该基因共表达的基因集合。
TISCH2数据库对全球用户免费开放,其中整合的数据资源将有助于肿瘤生物学家研究肿瘤微环境中的基因调控和免疫信号,并为发现新的药物靶点提供方向。TISCH项目团队将对该数据库进行定期的更新与维护,及时吸纳新发表的肿瘤病人样本的单细胞转录组数据,建立肿瘤免疫微环境大数据平台,为癌症研究、肿瘤免疫研究提供更好的服务。
同济大学生命科学与技术学院王晨飞研究员和华西口腔医院李太文副研究员为该论文共同通讯作者,同济大学博士研究生韩雅、王玉婷、董鑫为共同第一作者。数据库的建立得到了国家自然科学基金和上海市科委的基金项目支持。
参考文献:
[1] Marangoni,F et al. Expansion of tumor-associated Treg cells upon disruption of a CTLA-4-dependent feedback loop. Cell. 184,3998-4015 (2021)
[2] Sun D, et al. TISCH: a comprehensive web resource enabling interactive single-cell transcriptome visualization of tumor microenvironment. Nucleic Acid Res. 49 D900-908 (2021)
[3] Wang C, et al. Integrative analyses of single-cell transcriptome and regulome using MAES
TRO. Genome Biol. 21:198 (2020)
[4] Puram S, et al. Single-Cell Transcriptomic Analysis of Primary and Metastatic Tumor Ecosystems in Head and Neck Cancer. Cell. 171(7):1611-1624 (2017)
[5] Qin Q, et al. Lisa: inferring transcriptional regulators through integrative modeling of public chromatin accessibility and ChIP-seq data. Genome Biol. 21:32 (2020)
[6] Jin S, et al. Inference and analysis of cell-cell communication using CellChat. Nat Commun. 12,1088(2021)