你们好,Watson和Crick,我是Hoogsteen,我们来折叠吧
近年来,DNA纳米结构领域已在诸如环境监测、生物计算、智能材料等众多领域显现出可观的应用前景。凭借优异的可编码性,DNA分子通过精确的碱基序列互补配对得以实现复杂的高阶组装,从而形成具有特殊形貌、既定功能的纳米结构。迄今为止,绝大多数DNA纳米结构的研究皆是依赖于对单链DNA分子之间组装行为的操控,罕有文献记载过将自然界更为常见的双链DNA作为构筑单元进行纳米结构的设计和构建。这种对单链DNA的高度依赖性在很大程度上限制了DNA纳米结构在细胞内的应用空间。鉴于此,我们开发了一种全新的DNA纳米结构设计策略,该策略可以将双链DNA作为构筑单元从而使其进行组装形成复杂的纳米结构。
基于前期对DNA三螺旋的诸多探索,我们对三螺旋的结构和性质有了不断深入的了解,相关设计理念和设计经验也得到了不断的积累和丰富。这里,我们根据DNA三螺旋的特点,尝试将其应用于双链DNA的自组装。DNA三螺旋是在普通的Watson-Crick双螺旋的基础上,通过第三条链(TFO)以Hoogsteen或反式Hoogsteen碱基配对的方式插入到双链DNA的大沟之中形成的。受此结构特性启发,我们对TFO链进行了重新设计,使每条TFO链包含两部分,每部分都可以独立地识别一条较长的双链DNA骨架上的特定位点。在众多TFO链的辅助下,该双链DNA骨架通过其上众多结合位点之间复杂有序的连接网络进行折叠组装,形成符合预期设计的纳米结构。通过这种组装策略,我们成功的设计了多种多样的DNA纳米结构,这些纳米结构的正确组装也进一步验证了此策略的可行性和可靠性。
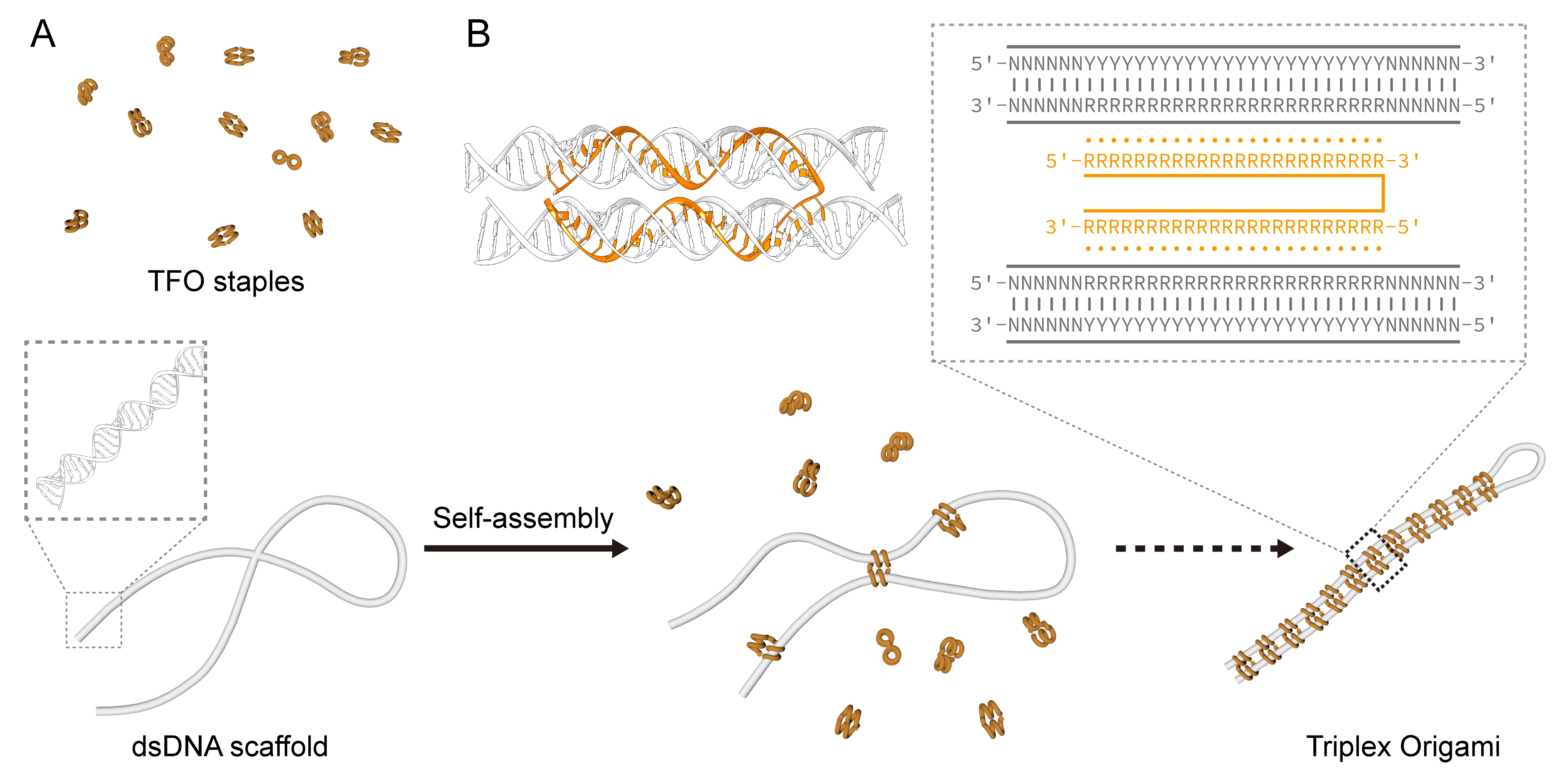
图 1. 利用三螺旋结构元素指导双链DNA进行折叠组装。
(A)组装过程示意图。(B)三螺旋结构形成位点以及TFO链结构示意图
凭借着此策略对双链DNA构象的可控性调节,我们接下来又初步探索了双链DNA构象调节在转录活动中可能发挥的作用。我们将目的基因前端序进行折叠形成特定的构象,使其抑制转录元件与启动子之间的识别和结合从而抑制相关基因的转录水平。并通过模块化的组装设计,确定了构象折叠程度与转录水平的关联。本研究建立了新的组装策略,拓展了核酸纳米结构的设计空间,丰富了DNA纳米结构的应用前景,对今后DNA纳米结构领域的发展具有重要的意义。
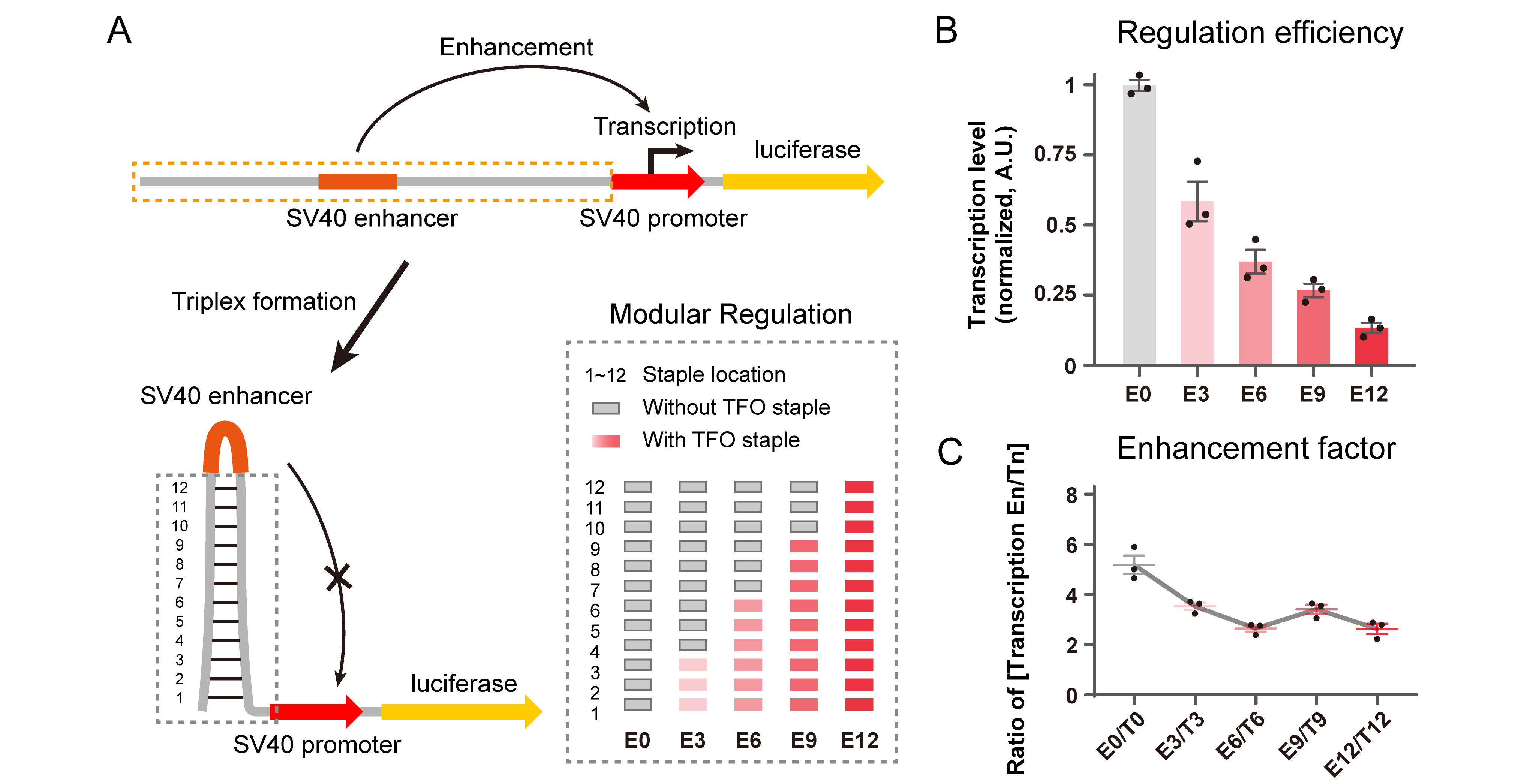
图 2. 基于对双链DNA构象的调控实现对相关基因转录水平的可控调节。 利用纳米结构的空间位阻,影响相关转录因子与启动子之间的结合从而控制转录水平。
该研究成果由清华大学生命学院魏迪明分子设计课题组(MADlab)和清华大学免疫所、医学院曾文文课题组合作完成,论文题为“基于三螺旋结构实现双链DNA的可控组装”(Custom folding of double-stranded DNA directed by triplex formation),于2023年3月21日在线发表于《化学》(Chem)期刊。清华大学生命科学学院已毕业的2017级博士生张天晴和医学院2017级博士生钱鑫民为本文的共同第一作者。其中张天晴和清华大学生命学院魏迪明副教授、清华大学免疫所曾文文副教授共同为本文的通讯作者。非常值得一提的,本文是张天晴自主设计和构架的DNA三螺旋三部曲的收官之作,其在研究生阶段就展现出了独立的探索精神和卓绝的执行力。该研究得到科技部、国家自然科学基金委、清华-北大生命科学联合中心等基金资助。
原文链接:
https://doi.org/10.1016/j.chempr.2023.02.018
相关论文:
https://doi.org/10.1021/jacs.1c07824
https://doi.org/10.1093/nar/gkac478