Nucleic Acids Research | 中山大学黄林团队发现一系列重要规律仅通过序列即能准确预测一种RNA motif的折叠与结构构象
【研究背景】
当前蛋白质结构预测发展迅猛且取得极大进展,然而RNA结构预测还存在很多困难,其中重要一点是RNA三维结构的重要组成RNA motif研究少,且从中提取的折叠与结构规律不足。
非编码RNA(ncRNA)与基因的表达调控是当前生命科学最活跃的前沿研究之一。为了理解ncRNA功能,研究RNA的折叠过程与三维结构就显得尤为重要。
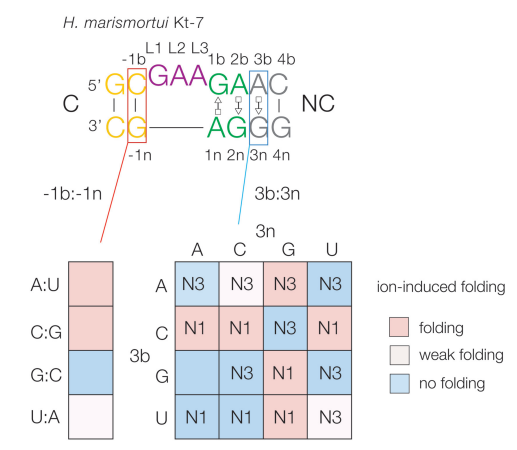
K-转角(k-turn)是一类广泛存在于包括核糖体、核糖开关、snoRNA以及lncRNA等的关键结构元件RNA motif,在体内发挥重要作用(图一)。
【主要发现】
在本研究中,黄林课题组发现一系列重要的规律,基于这些规律仅通过序列即能准确预测k-转角在溶液状态下能否正确折叠以及原子尺度精确的结构构象(图一)。将相关规律应用于Breaker 实验室发现的 224 种结构化RNA中,从而鉴定出四个可能的 k-转角(图二),根据相关规则预测其折叠和三维结构。
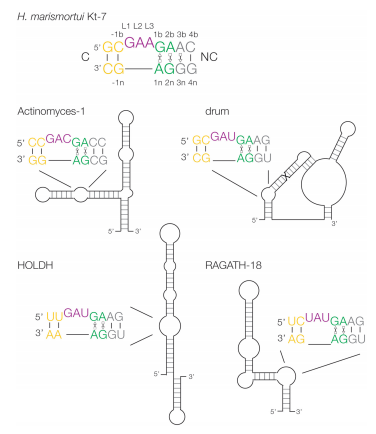
通过荧光共振能量转移(FRET)分析四个k-转角折叠的离子依赖性,并用X-射线晶体学确定了其中两个k-转角的构象,发现实验数据完全符合预测的折叠和三维结构特性。总之,我们的预测规则是可靠而准确的,可以很好的应用于RNA结构预测中。
【研究意义】
本研究是整合各方资源系统研究RNA motif的典型代表,公认为研究最为透彻RNA motif,仅通过序列即能准确预测k-转角的折叠与结构构象。因此,为推进与提高以序列为基础预测RNA分子结构的计算方法提供了理论与实践基础。
论文链接: https://doi.org/10.1093/nar/gkab333
通讯作者:黄林等
第一作者:黄林;李梦筱等