服务热线
178 0020 3020
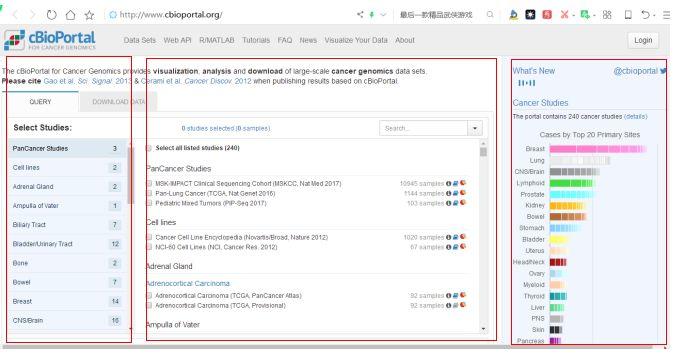
要问分析TCGA数据库大家最讨厌什么?就小编而言,当初学习TCGA数据库时候最讨厌用R软件分析时候代码的报错!这个问题估计是相当苦恼的,今天,小编将为大家介绍一个无需代码,并且免费使用的TCGA子数据库——cBioportal网站。毋庸置疑,这是一个科研汪们集万千宠爱的网站。 首先,我们进入网站了解一下。官网网址为http://www.cbioportal.org/。进入官网后,主页分为以下三个区域,左侧的为选择研究的区域(按器官对各肿瘤分类),中间是下载研究数据的区域,右侧区域是对各研究数据的一个总结及该癌种的临床信息。
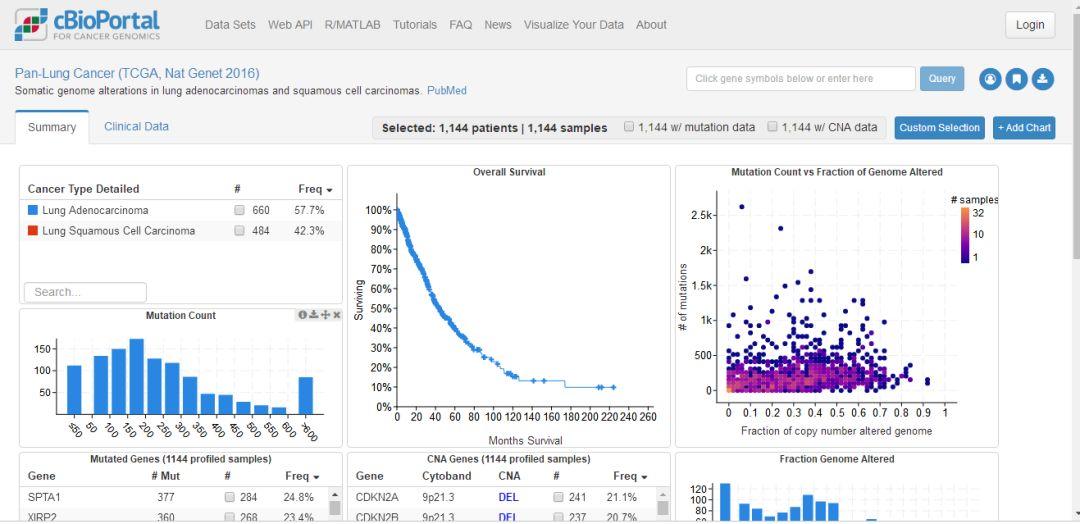
 我们先点击右侧区域的“Lung”了解一下,可以看到这是对肺癌所有研究数据的一个总结。
我们先点击右侧区域的“Lung”了解一下,可以看到这是对肺癌所有研究数据的一个总结。
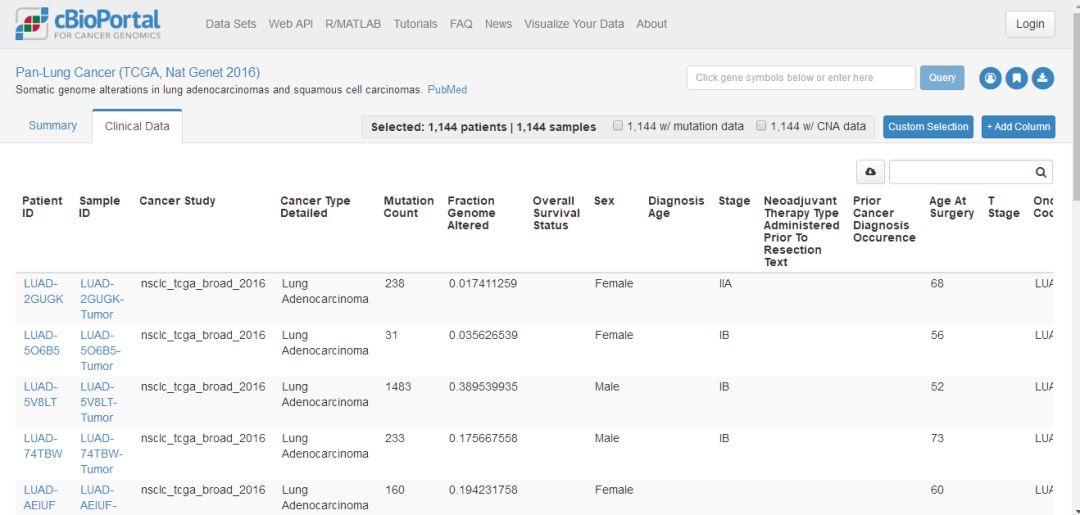
 点击“Clinical Data”,可以查看肺癌患者的临床信息。
点击“Clinical Data”,可以查看肺癌患者的临床信息。
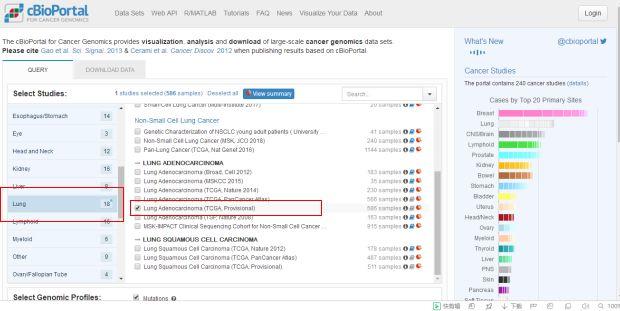
 小编主要研究的是胸部肿瘤,所以今天就以TCGA数据库肺腺癌数据来举例分析。首先,我们找到左侧的“Lung”,点击选项,再选择一个肺腺癌的数据集。
小编主要研究的是胸部肿瘤,所以今天就以TCGA数据库肺腺癌数据来举例分析。首先,我们找到左侧的“Lung”,点击选项,再选择一个肺腺癌的数据集。
 下拉页面,选择数据类型,包括突变数据、拷贝变异数据、mRNA表达数据、蛋白磷酸化数据,本帖我们选择分析这四种数据。接下来选择基因,选择TGF-β通路的43个基因,当然,也可以选择自己已经获得的Hub基因或目标基因,手动输入到基因输入框中。输入成功后,数据库会自动判断输入的基因是否有效,绿色代表输入成功,最后点击提交。
下拉页面,选择数据类型,包括突变数据、拷贝变异数据、mRNA表达数据、蛋白磷酸化数据,本帖我们选择分析这四种数据。接下来选择基因,选择TGF-β通路的43个基因,当然,也可以选择自己已经获得的Hub基因或目标基因,手动输入到基因输入框中。输入成功后,数据库会自动判断输入的基因是否有效,绿色代表输入成功,最后点击提交。
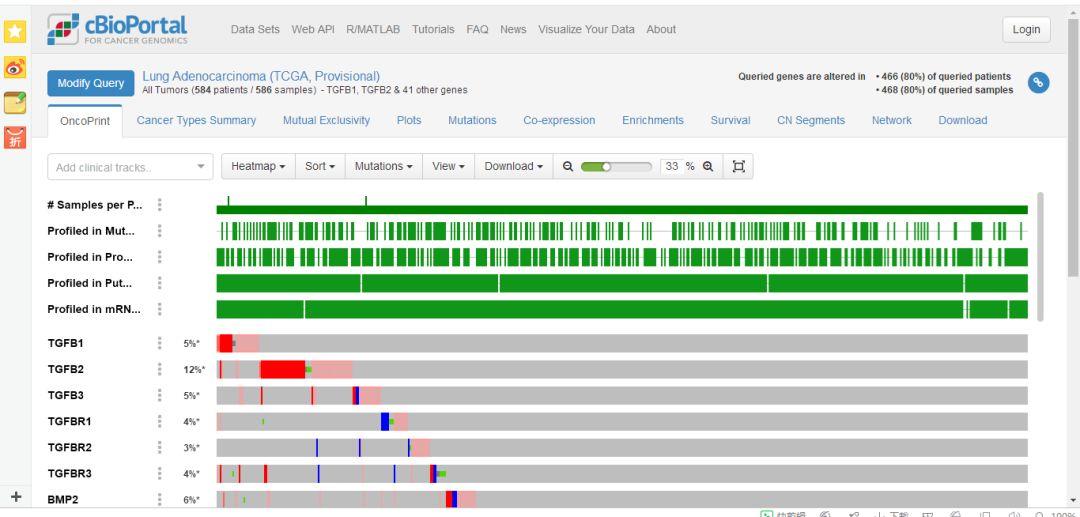
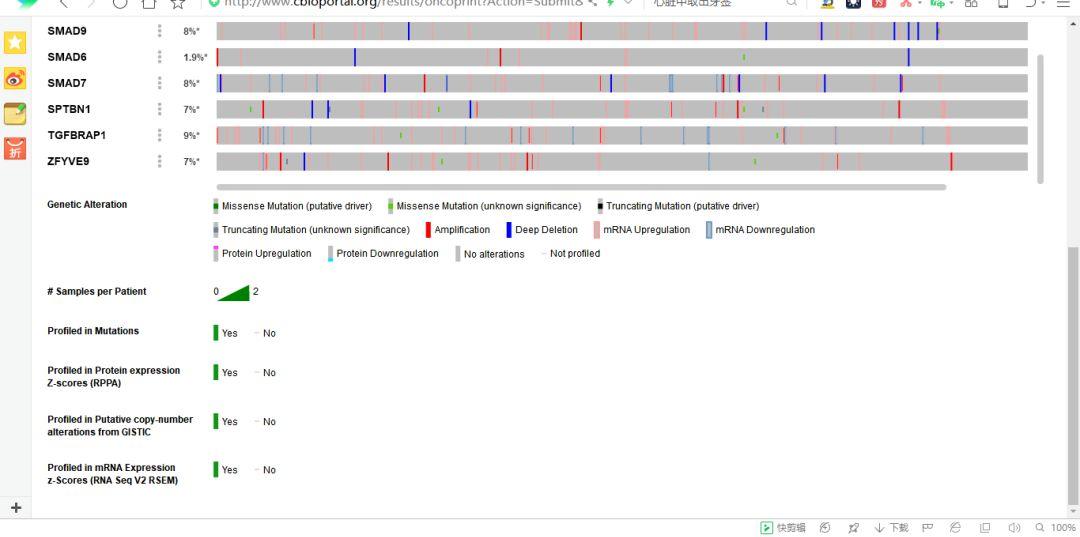
 弹出的第一个页面,显示的各个基因对应的总体样本的分布情况,下拉页面,图形中各个颜色的含义都做了备注。
弹出的第一个页面,显示的各个基因对应的总体样本的分布情况,下拉页面,图形中各个颜色的含义都做了备注。
 在这个页面的右上角,提供了一个热图选项,小编试着将其点开。
在这个页面的右上角,提供了一个热图选项,小编试着将其点开。
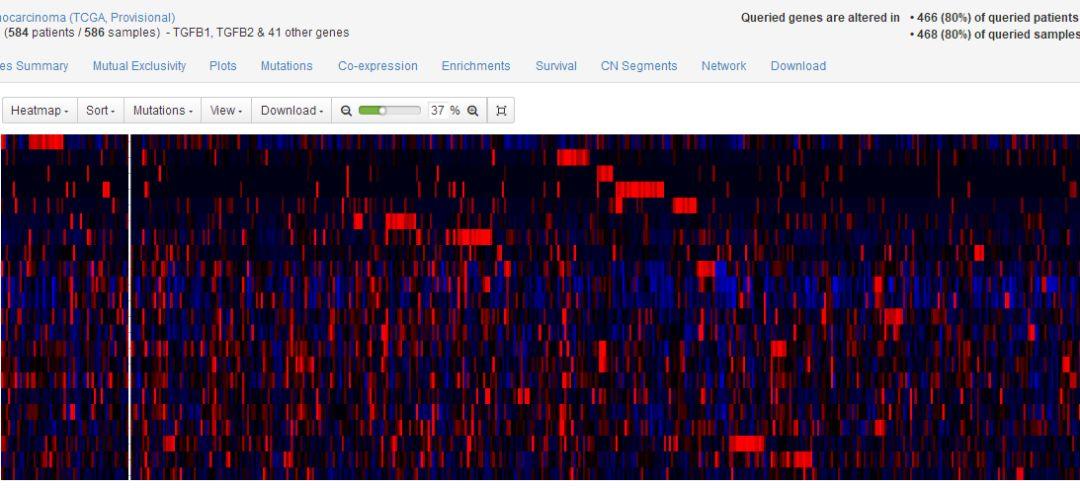
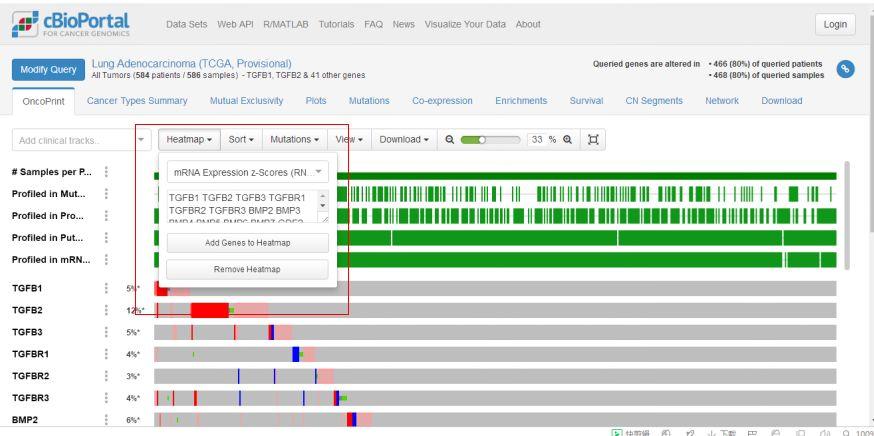 点击“Add Genes to Heatmap”,得到一幅热图,这热图看起来怪怪的,不过也能凑合着看了。
点击“Add Genes to Heatmap”,得到一幅热图,这热图看起来怪怪的,不过也能凑合着看了。
 第二个页面是肺腺癌各数据类型的分布,包括突变、扩增、高度缺失、mRNA高表达、mRNA低表达、蛋白低表达、复杂改变。cBioPortal for Cancer Genomics提供大规模癌症基因组数据集的可视化和分析。
第二个页面是肺腺癌各数据类型的分布,包括突变、扩增、高度缺失、mRNA高表达、mRNA低表达、蛋白低表达、复杂改变。cBioPortal for Cancer Genomics提供大规模癌症基因组数据集的可视化和分析。
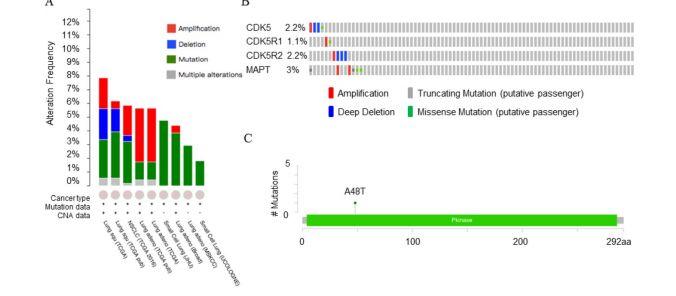
 有文献使用cBioPortal数据库中的8个肺癌数据集研究了肺癌中的CDK5的基因组变化,其改变包括扩增,缺失,突变和复杂改变,其中6个数据集中突变比重最大,2个数据集中扩增的比重最大(图A)。(获取文献方式,请看文末)
有文献使用cBioPortal数据库中的8个肺癌数据集研究了肺癌中的CDK5的基因组变化,其改变包括扩增,缺失,突变和复杂改变,其中6个数据集中突变比重最大,2个数据集中扩增的比重最大(图A)。(获取文献方式,请看文末)
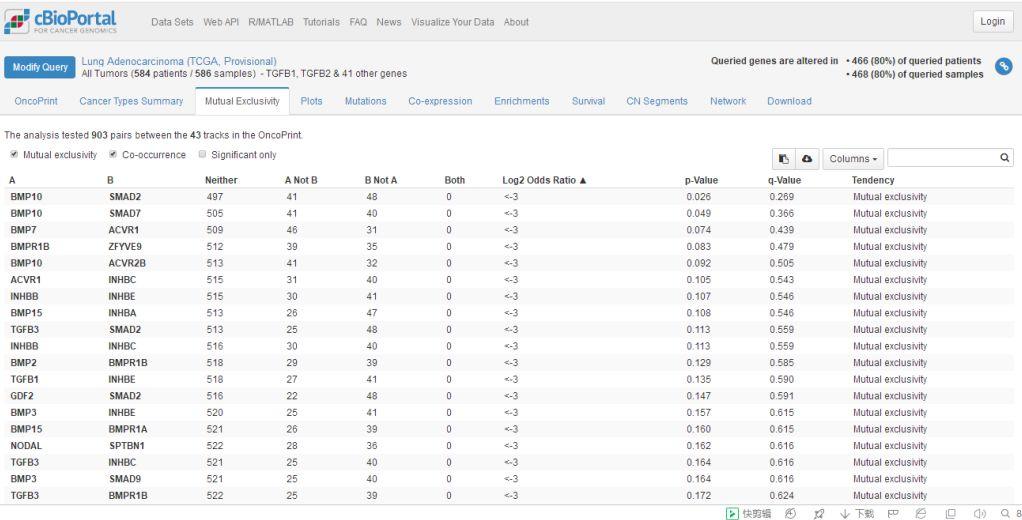
 第三个页面,显示的是两个基因与肿瘤的相关性,假设A基因与肿瘤相关,那么B基因与肿瘤相关性就小一些,挺拗口的!我们可以通过Log2OR大小来判断这种关系,如果Log2OR越小,则说明A基因与肿瘤更相关,而B基因与肿瘤相关性则越小。
第三个页面,显示的是两个基因与肿瘤的相关性,假设A基因与肿瘤相关,那么B基因与肿瘤相关性就小一些,挺拗口的!我们可以通过Log2OR大小来判断这种关系,如果Log2OR越小,则说明A基因与肿瘤更相关,而B基因与肿瘤相关性则越小。
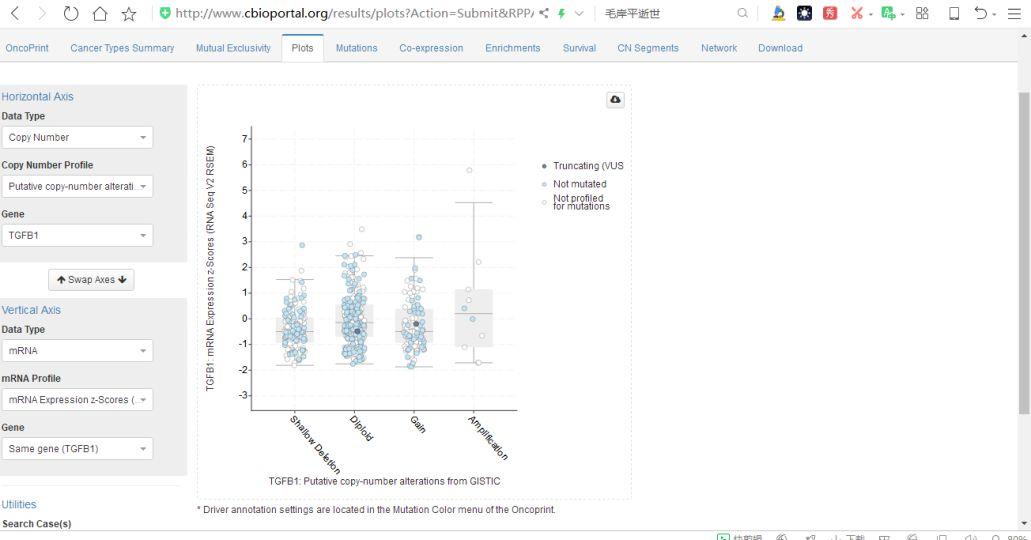
 第四个页面是一幅基因分析的离散图,右侧的图的意思是肺腺癌中TGFB1基因与拷贝数变化的散点图。左侧的菜单中的基因、数据类型都可以更改,根据需要,选择不同的数据来作图。
第四个页面是一幅基因分析的离散图,右侧的图的意思是肺腺癌中TGFB1基因与拷贝数变化的散点图。左侧的菜单中的基因、数据类型都可以更改,根据需要,选择不同的数据来作图。
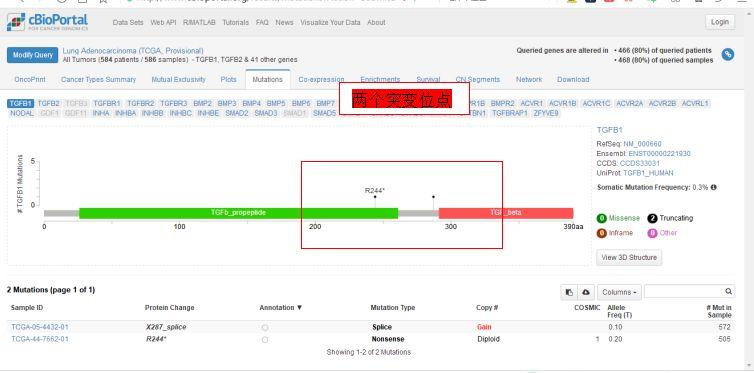
 第五个页面是查看突变的位置及类型的,可以看出突变位点有两个,分别是X287-splice(可变剪接位点)和R244*(无义突变位点)。
第五个页面是查看突变的位置及类型的,可以看出突变位点有两个,分别是X287-splice(可变剪接位点)和R244*(无义突变位点)。
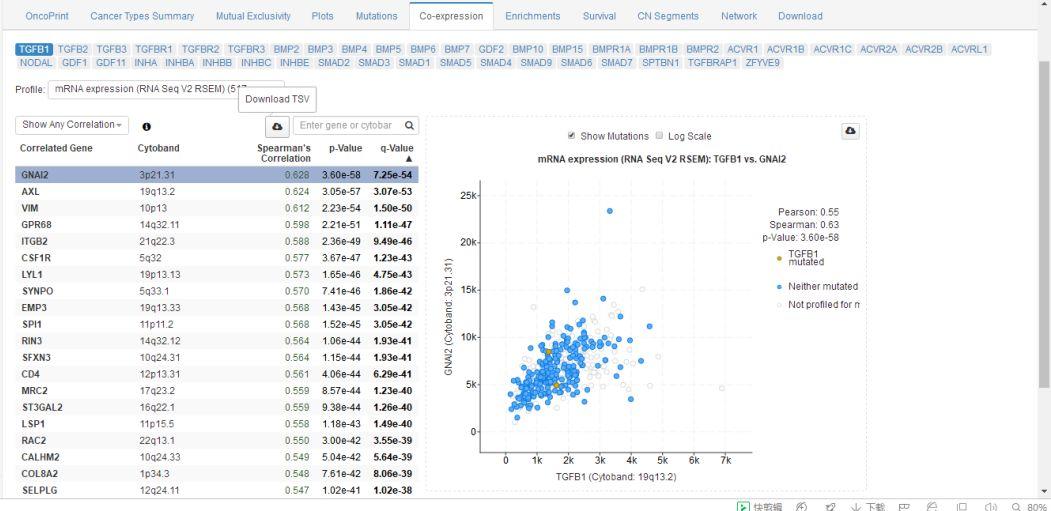
 第六个页面显示的是共表达情况,左侧是与TGF-β1相关的共表达基因,右侧是某个基因与TGF-β1共表达的关系图,共表达图直接可以免费下载,用起来非常方便。
第六个页面显示的是共表达情况,左侧是与TGF-β1相关的共表达基因,右侧是某个基因与TGF-β1共表达的关系图,共表达图直接可以免费下载,用起来非常方便。
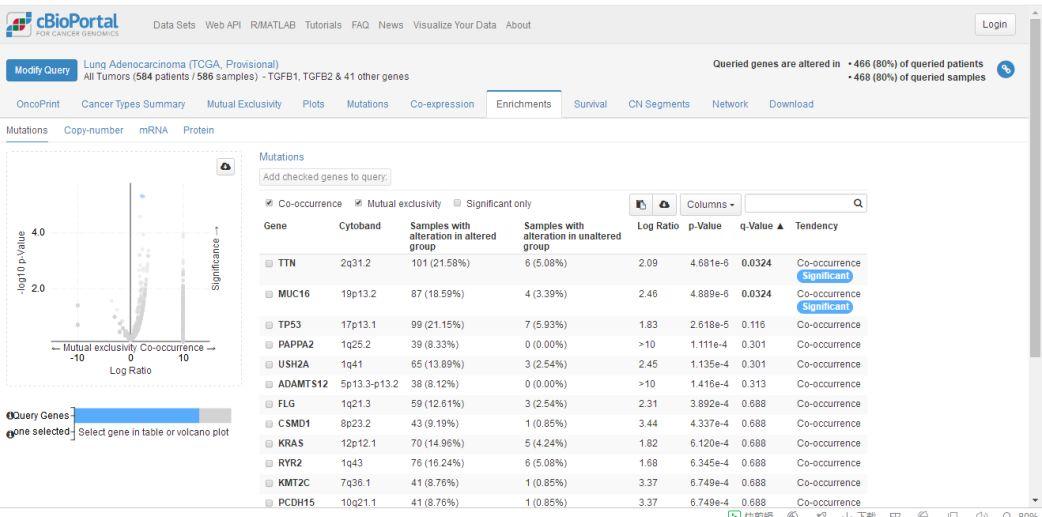
 第七个页面是富集分析的情况。左侧是这43个基因突变数据的一幅火山图,同样也可以查看拷贝数、mRNA表达、蛋白数据的火山图。
第七个页面是富集分析的情况。左侧是这43个基因突变数据的一幅火山图,同样也可以查看拷贝数、mRNA表达、蛋白数据的火山图。
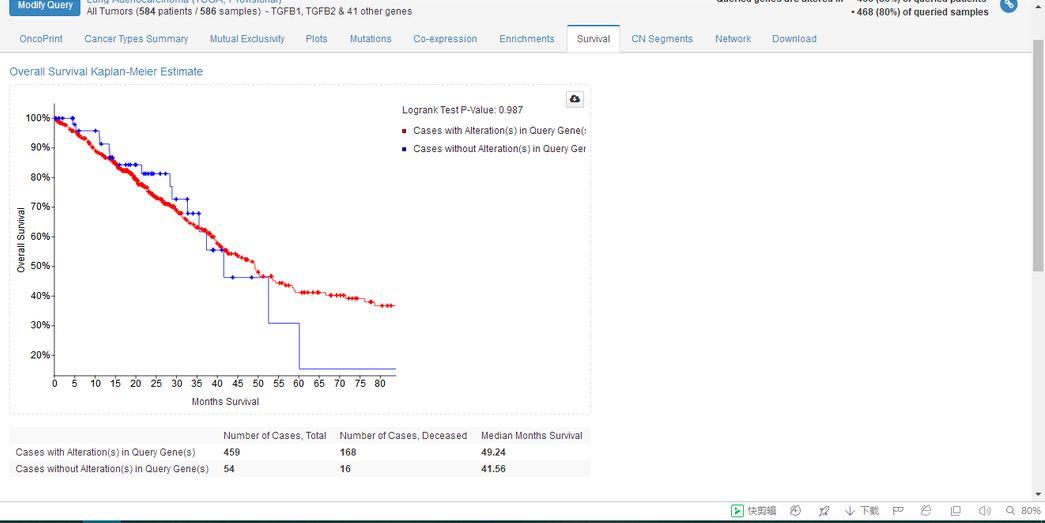
 第八个页面是生存分析,第一幅图查看的是总生存期的生存曲线。
第八个页面是生存分析,第一幅图查看的是总生存期的生存曲线。
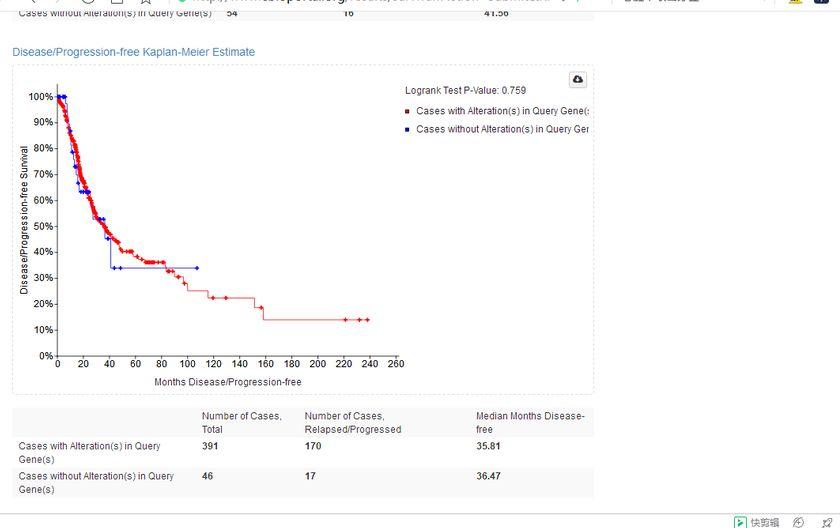
 下拉页面,查看无进展生存期的生存曲线。
下拉页面,查看无进展生存期的生存曲线。
 第九个页面是拷贝数的具体信息,点击Download下载拷贝变异数据。
第九个页面是拷贝数的具体信息,点击Download下载拷贝变异数据。
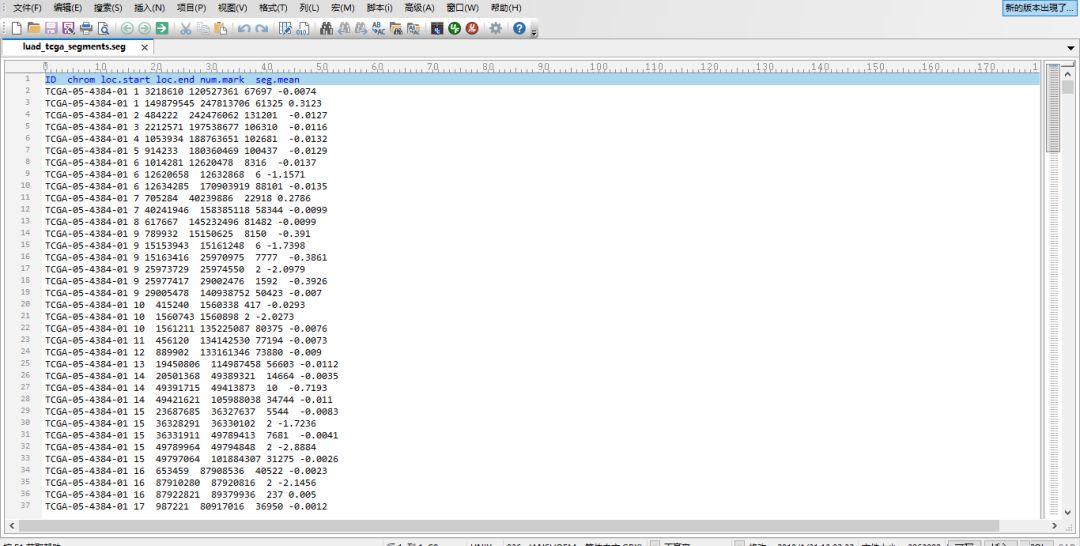
 打开文档,可以查看基因变异的位置。
打开文档,可以查看基因变异的位置。
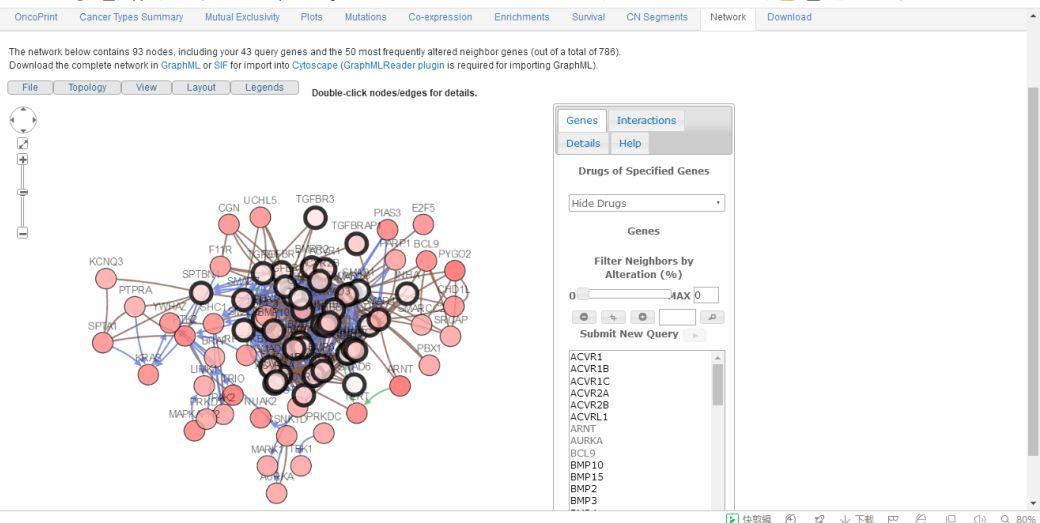
 第十个页面是蛋白互作网络图(PPI),这个不用多说,点击页面加以调整即可在文章中使用。
第十个页面是蛋白互作网络图(PPI),这个不用多说,点击页面加以调整即可在文章中使用。
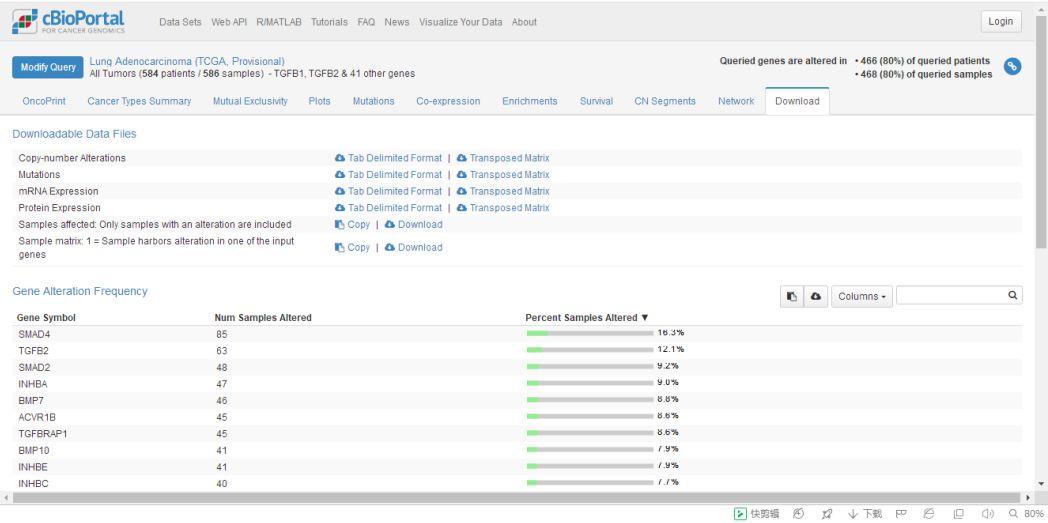
 最后一个页面,是数据下载的页面,各类型数据的原始数据以及矩阵均可以在这个页面进行下载。
最后一个页面,是数据下载的页面,各类型数据的原始数据以及矩阵均可以在这个页面进行下载。
 比如,下载一个肺腺癌基因表达的矩阵。
比如,下载一个肺腺癌基因表达的矩阵。
 看着还不错吧,是不是惊讶这样也能分析TCGA数据?cBioportal网站不得不说是小伙伴们的福利,操作非常简单,数据免费分析,重点只是对数据的解读。学了本帖,小伙伴们赶紧行动起来吧!
看着还不错吧,是不是惊讶这样也能分析TCGA数据?cBioportal网站不得不说是小伙伴们的福利,操作非常简单,数据免费分析,重点只是对数据的解读。学了本帖,小伙伴们赶紧行动起来吧!
附件