服务热线
178 0020 3020
之前在介绍 GEO 的时候,提到过里面包括了很多其他研究人员发表文章的时候使用的测序数据。其中就包括circRNA 测序的数据。但是对于 circRNA 测序的数据的话,如果是用 GEO2R 分析相关的芯片,就需要面临一个芯片ID-circRNA注释的问题。这个还需要一些功夫去研究。如果是研究肿瘤的话,那就幸运了,今天就给大家介绍一个把 和肿瘤有关的 circRNA 测序数据差异分析的数据库:circExp (http://soft.bioinfo-minzhao.org/circexp/index.html#faq)

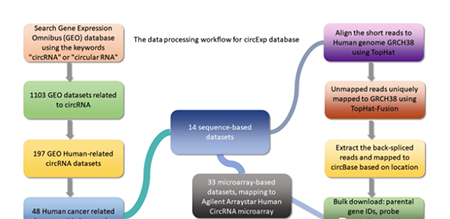
circExp主要使用的还是 GEO 里面的数据。通过检索circRNA 相关的数据。然后再筛选和肿瘤有关的 circRNA 数据。最后对数据进行统一处理。
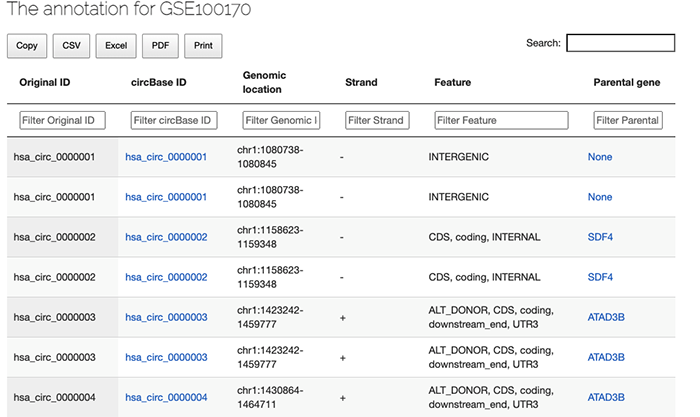
在 circExpr 当中,我们只能查看每个数据集注释信息。并没有每一个数据集的差异分析结果。
但是。在Download 里面就可以下载作者检索到的那些数据集的差异分析的结果。
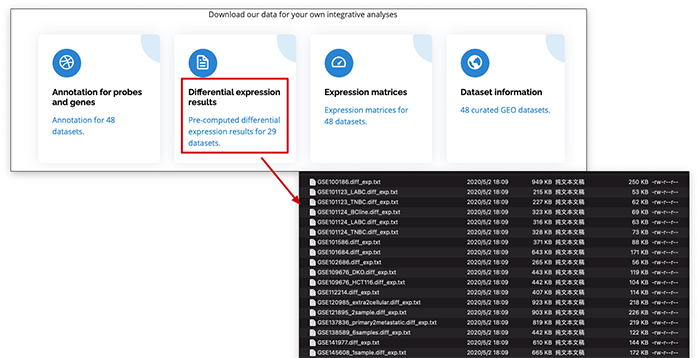
所以可以直接下载所有的结果即可得到肿瘤相关的 circRNA 的差异表达结果了。
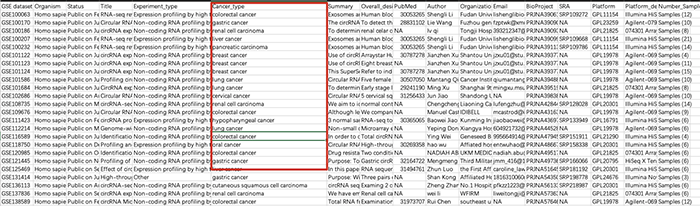
至于说每个数据集是什么癌,可以下载DataSet Information 即可查看。
一个已经公开并且已经发表了的数据库其中一些功能非常的不完善的。首先在其基因注释功能的检索方面 居然有一些数据集的注释结果 NA 。但是这个基因结构明显在 circBase 里面是有具体的 ID的。
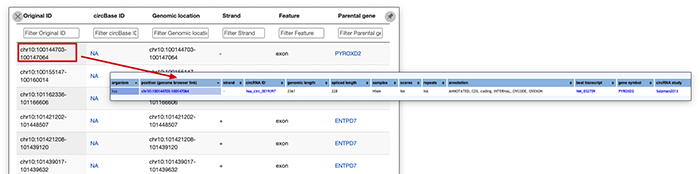
另外在数据库功能方面。在下载的里面有差异表达的结果,但是在数据库使用里面居然没有这个功能。明显要是不全面的。
附件