http://www.ribocirc.com 大家使用的时候也不要忘记引用参考文献
Li, H., Xie, M., Wang, Y. et al. riboCIRC: a comprehensive database of translatable circRNAs. Genome Biol 22, 79 (2021). https://doi.org/10.1186/s13059-021-02300-7
一、riboCIRC数据库基本介绍
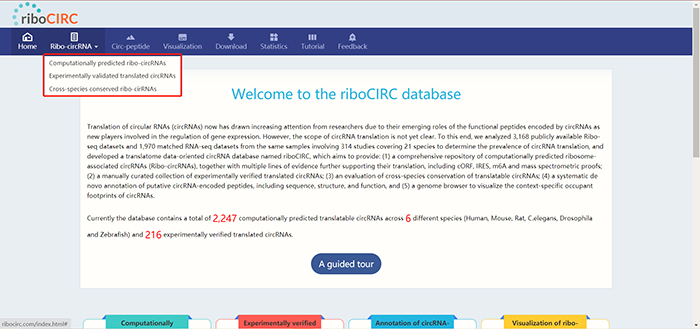
riboCIRC数据库,研究的也是circRNA编码多肽,就机制而言同TransCirc数据库大同小异。所以我仔细想了想,什么叫ORF,什么叫IRES,什么是circRNA编码蛋白的机制,以及包括m6A修饰这些,在之前的推文中都悉数介绍过了,所以大家再了解一下这个数据库的基本构建,基本上就掌握的八九不离十了。大家在使用的时候可以跟Transcirc数据库进行对比思考,以加深理解。如Transcirc数据库对预测circRNA是否可以编码蛋白进行了证据等级评分,从多个方面来预测circRNA编码蛋白的可能性;而riboCIRC数据库则简单粗暴地直接使用了Ribo-seq数据集进行分析,更加可信。riboCIRC数据库分析了3168个Ribo-seq公共数据集以及1970个相匹配的RNA-seq数据集,包括了314项研究,21个物种的信息,最终完成了一个以翻译组学为导向的circRNA数据库。数据库目前包含了6个不同物种(人,小鼠,大鼠,线虫,果蝇,斑马鱼)的2247条预测可以进行翻译的circRNA以及216条经实验验证过可以翻译的circRNA,原理如下图所示: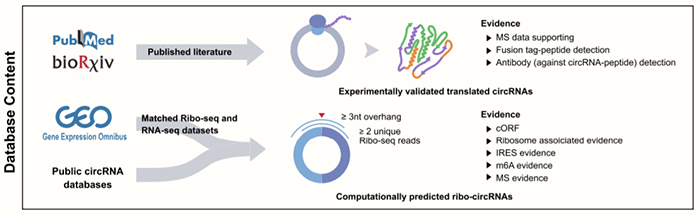 同时,riboCIRC数据库相比Transcirc数据库更优秀的一点就是它所收录的信息更为丰富,还包括了circRNA的可视化,引物设计,以及所编码多肽的理化性质,亚细胞定位等等,如下图所示:
同时,riboCIRC数据库相比Transcirc数据库更优秀的一点就是它所收录的信息更为丰富,还包括了circRNA的可视化,引物设计,以及所编码多肽的理化性质,亚细胞定位等等,如下图所示: 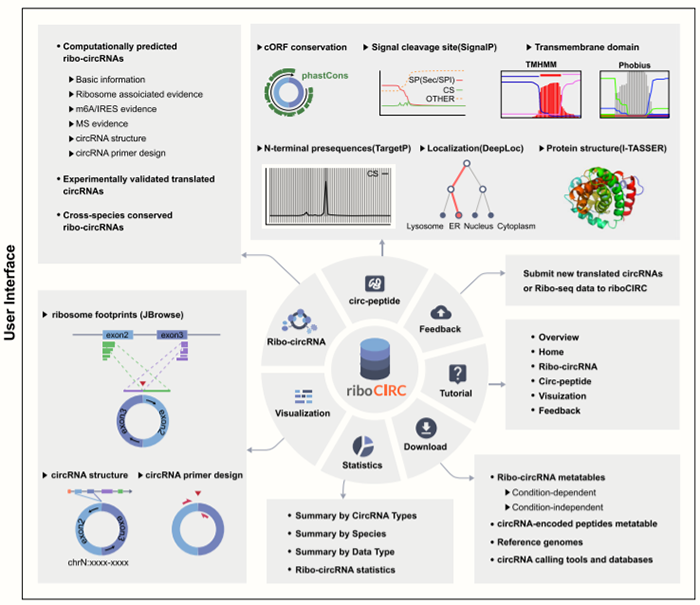
二、riboCIRC数据库实操演示
输入网址http://www.ribocirc.com,进入数据库主页面: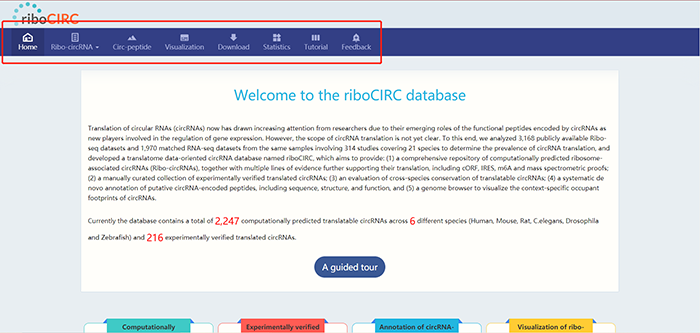 按照以往习惯,我们还是先来看看菜单栏的内容。riboCIRC数据库菜单栏包括“Home”(当前主页),“Ribo-circRNA”(包括算法预测的ribo-circRNA,实验验证的可翻译circRNA以及物种保守性circRNA),“Circ peptide”(基于生物信息学预测提供了circRNA所编码小肽的序列,结构以及功能),“Visualization”(基因组浏览器可视化),“Download”(下载),“Statistics”(数据统计),“Tutorial”(使用指南),以及“Feedback”(反馈)。 页面继续下拉,可以看到上述几个功能的快捷入口,用户可以直接点击相应title进入链接,或者同上述点击菜单栏即可:
按照以往习惯,我们还是先来看看菜单栏的内容。riboCIRC数据库菜单栏包括“Home”(当前主页),“Ribo-circRNA”(包括算法预测的ribo-circRNA,实验验证的可翻译circRNA以及物种保守性circRNA),“Circ peptide”(基于生物信息学预测提供了circRNA所编码小肽的序列,结构以及功能),“Visualization”(基因组浏览器可视化),“Download”(下载),“Statistics”(数据统计),“Tutorial”(使用指南),以及“Feedback”(反馈)。 页面继续下拉,可以看到上述几个功能的快捷入口,用户可以直接点击相应title进入链接,或者同上述点击菜单栏即可: 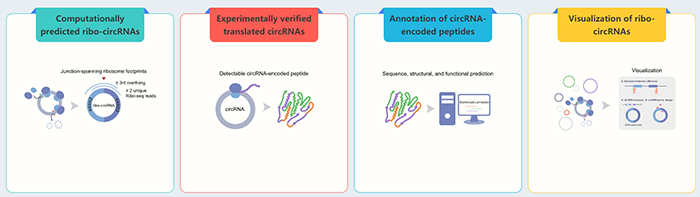 页面最下面则是该数据库的一些基本信息:
页面最下面则是该数据库的一些基本信息:  我们接下来介绍一下数据库最重要的几个功能:
我们接下来介绍一下数据库最重要的几个功能:
1
“Ribo-circRNA”----检索可编码的circRNA
菜单栏处点击“Ribo-circRNA”,可以看到进一步分为了三个子功能:“Computationally predicted ribo-circRNAs”,“Experimentally validated translated circRNAs”,“Cross-species conserved ribo-circRNAs”:
 1. “Computationally predicted ribo-circRNAs” 点击第一个“Computationally predicted ribo-circRNAs”,跳转到如下页面:
1. “Computationally predicted ribo-circRNAs” 点击第一个“Computationally predicted ribo-circRNAs”,跳转到如下页面: 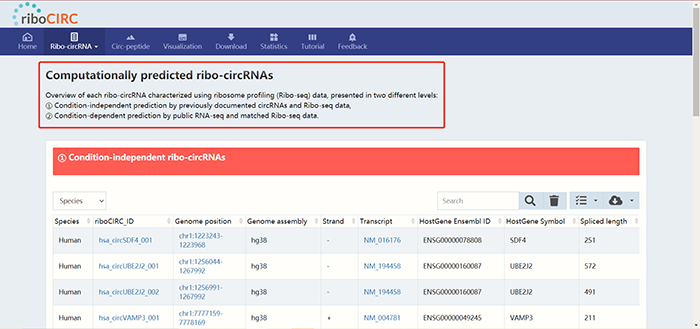 在这里我们先做一个前情提要,riboCIRC数据库主要收录的是可以编码的circRNA,也就是同核糖体有紧密联系的circRNA。同前文所述,开发者使用了两种类型的数据集进行分析,一类是Ribo-seq,另一类则是相应的RNA-seq,通过比对以及使用circRNA鉴定的算法来明确核糖体上结合的circRNA有哪些。其实还有一种思路,就是不用RNA-seq的数据,直接使用其他数据库中所收录的已经报道过的circRNA,这样就省去了进一步去鉴定circRNA的麻烦。因此在这里,riboCIRC数据库根据这个circRNA是否已有文献报道/数据库收录同样分为了两部分:Condition-independent ribo-circRNAs以及Condition-dependent ribo-circRNAs:
在这里我们先做一个前情提要,riboCIRC数据库主要收录的是可以编码的circRNA,也就是同核糖体有紧密联系的circRNA。同前文所述,开发者使用了两种类型的数据集进行分析,一类是Ribo-seq,另一类则是相应的RNA-seq,通过比对以及使用circRNA鉴定的算法来明确核糖体上结合的circRNA有哪些。其实还有一种思路,就是不用RNA-seq的数据,直接使用其他数据库中所收录的已经报道过的circRNA,这样就省去了进一步去鉴定circRNA的麻烦。因此在这里,riboCIRC数据库根据这个circRNA是否已有文献报道/数据库收录同样分为了两部分:Condition-independent ribo-circRNAs以及Condition-dependent ribo-circRNAs: 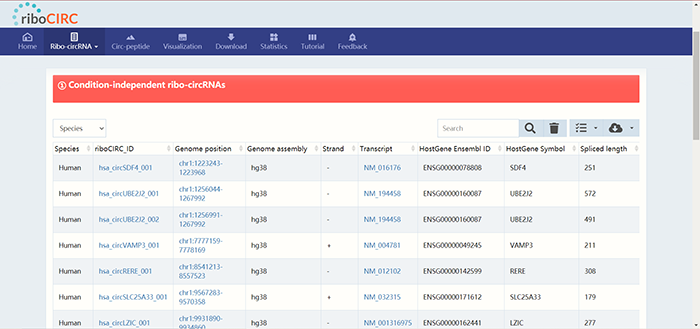
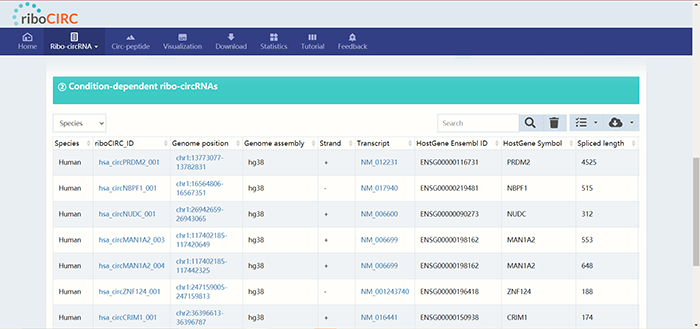 首先以“Condition-independent ribo-circRNAs”为例,表格从左到右依次展示了物种,riboCIRC的ID,染色体位置,参考基因组版本,正/反义链,最佳转录本,宿主基因的Ensemble ID,宿主基因的Gene Symbol,全长,参考数据集,以及支持编码的证据。表格还可以左右滑动进行查看,大家注意一下哦~点击“Genome position”,“Transcript”下方的超链接可以分别链接到基因组浏览器以及NCBI页面进一步查看。右上方检索框中可以输入circRNA ID进一步检索。
首先以“Condition-independent ribo-circRNAs”为例,表格从左到右依次展示了物种,riboCIRC的ID,染色体位置,参考基因组版本,正/反义链,最佳转录本,宿主基因的Ensemble ID,宿主基因的Gene Symbol,全长,参考数据集,以及支持编码的证据。表格还可以左右滑动进行查看,大家注意一下哦~点击“Genome position”,“Transcript”下方的超链接可以分别链接到基因组浏览器以及NCBI页面进一步查看。右上方检索框中可以输入circRNA ID进一步检索。 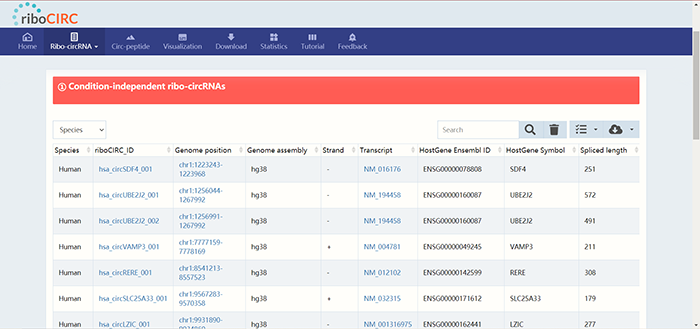
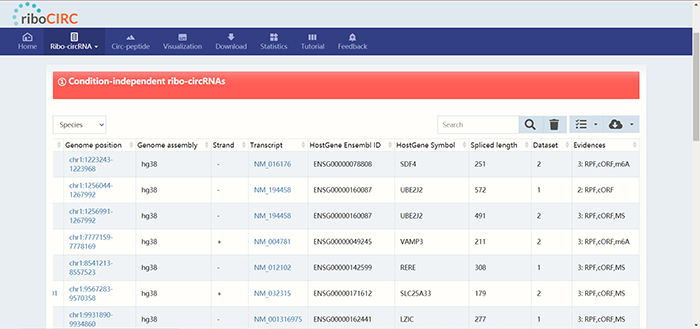 点击“riboCIRC_ID”下方的超链接可以查看该circRNA的详细内容。以第一条“hsa_circSDF4_001”为例,点击进入如下页面:
点击“riboCIRC_ID”下方的超链接可以查看该circRNA的详细内容。以第一条“hsa_circSDF4_001”为例,点击进入如下页面: 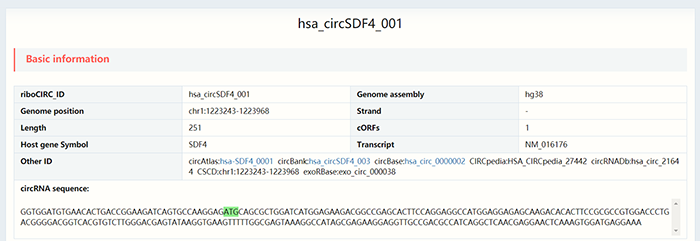 首先显示了该circRNA的基本信息,包括在riboCIRC数据库中的ID等。这里说明一下什么是cORFs,ORF的概念大家都知道,cORFs则是指ORF跨过了circRNA的反向剪接位点。由于“Condition-independent ribo-circRNAs”中的circRNA已有文献报道或被其他数据库收录过的,所以这里特意还加了一栏“Other ID”,提供了该circRNA在其他数据库中的ID,包括circAtlas数据库,circBank数据库,circBase数据库,CIRCpedia数据库,circRNADb数据库,CSCD数据库,exoRBase数据库等,全都已经被写成了推文,大家可以在挑圈联靠公众号查找。下方提供了circRNA的序列,其中起始密码子ATG用绿色标识进行突出。 页面继续下拉可以看到支持该circRNA编码的证据,包括使用的参考数据集,m6A位点,IRES位点,ORF以及MS证据。相关的理论知识大家可以参考circRNADb以及Transcirc数据库推文。
首先显示了该circRNA的基本信息,包括在riboCIRC数据库中的ID等。这里说明一下什么是cORFs,ORF的概念大家都知道,cORFs则是指ORF跨过了circRNA的反向剪接位点。由于“Condition-independent ribo-circRNAs”中的circRNA已有文献报道或被其他数据库收录过的,所以这里特意还加了一栏“Other ID”,提供了该circRNA在其他数据库中的ID,包括circAtlas数据库,circBank数据库,circBase数据库,CIRCpedia数据库,circRNADb数据库,CSCD数据库,exoRBase数据库等,全都已经被写成了推文,大家可以在挑圈联靠公众号查找。下方提供了circRNA的序列,其中起始密码子ATG用绿色标识进行突出。 页面继续下拉可以看到支持该circRNA编码的证据,包括使用的参考数据集,m6A位点,IRES位点,ORF以及MS证据。相关的理论知识大家可以参考circRNADb以及Transcirc数据库推文。 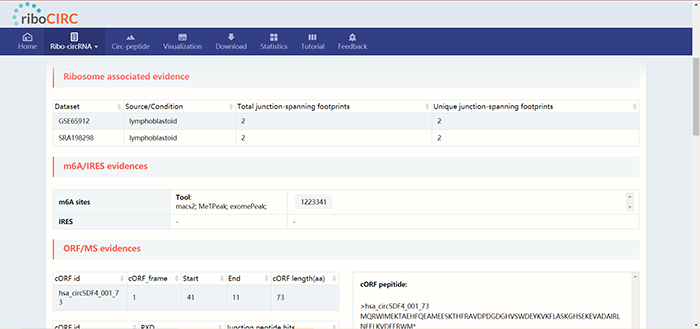 页面继续下拉可以查看circRNA的可视化结构,由几号外显子组成:
页面继续下拉可以查看circRNA的可视化结构,由几号外显子组成: 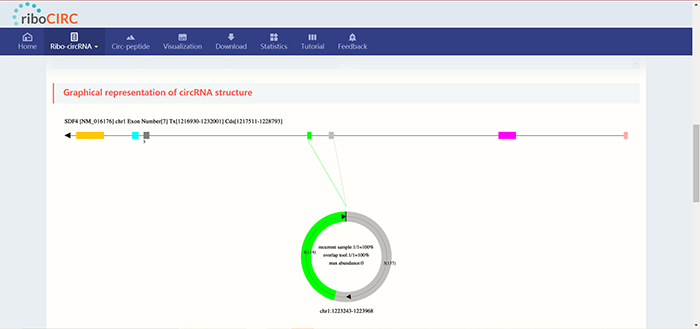 页面最后可以看到数据库联合circtools针对每一个circRNA所设计的引物,其中TM与GC%两个参数用红蓝色标识,数值越高,红色越深。
页面最后可以看到数据库联合circtools针对每一个circRNA所设计的引物,其中TM与GC%两个参数用红蓝色标识,数值越高,红色越深。 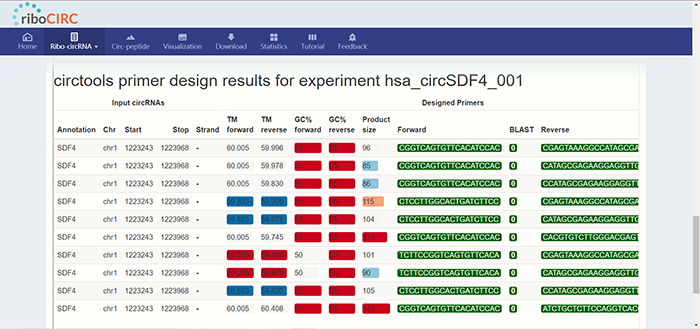 点击“Condition-dependent ribo-circRNAs”,结果页面同上,此处不再赘述。 2. “Experimentally validated translated circRNAs” 点击“Experimentally validated translated circRNAs”,跳转到如下界面:
点击“Condition-dependent ribo-circRNAs”,结果页面同上,此处不再赘述。 2. “Experimentally validated translated circRNAs” 点击“Experimentally validated translated circRNAs”,跳转到如下界面: 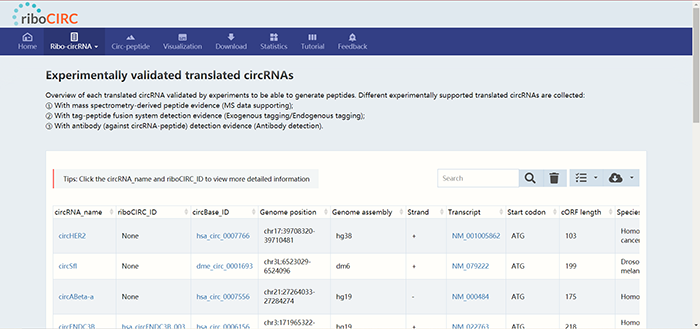
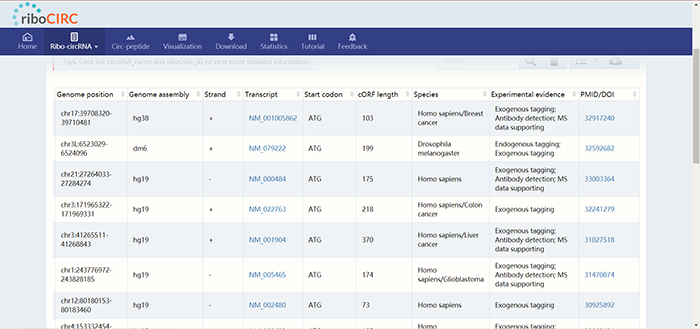 这里收录的均为经过实验验证,已有文献报道过可以编码的circRNA。实验证据包括质谱,亲和标签系统,抗体检测。点击“circRNA_name”下的超链接可以进一步查看所支持的文献信息:
这里收录的均为经过实验验证,已有文献报道过可以编码的circRNA。实验证据包括质谱,亲和标签系统,抗体检测。点击“circRNA_name”下的超链接可以进一步查看所支持的文献信息:  3. “Cross-species conserved ribo-cirRNAs” 点击“Cross-species conserved ribo-cirRNAs”可以查看在人和小鼠体内具有保守性的circRNA:
3. “Cross-species conserved ribo-cirRNAs” 点击“Cross-species conserved ribo-cirRNAs”可以查看在人和小鼠体内具有保守性的circRNA: 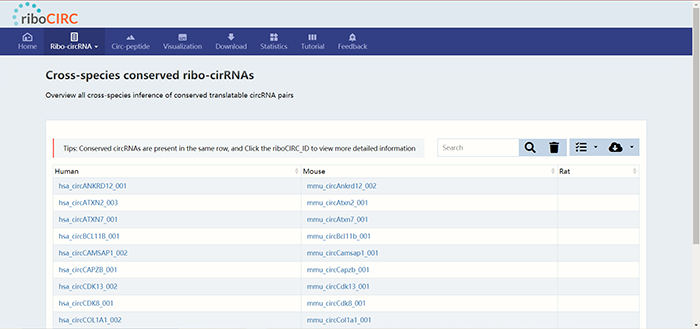
2
“Circ-peptide”----检索circRNA编码的小肽
点击“Circ-peptide”可以查看circRNA所编码小肽的基本信息。以hsa_circAAMDC_001_133为例,在检索框中输入后等待页面刷新:
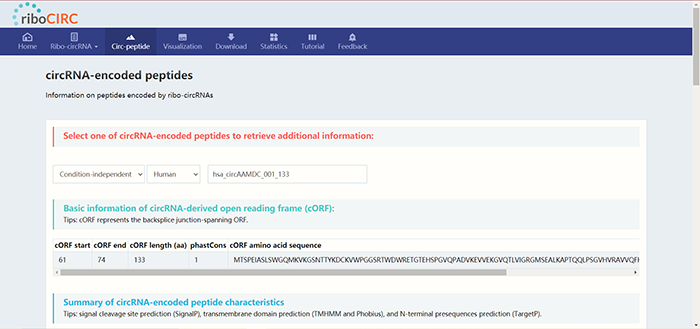 提供的内容包括cORF的起始和终止位置,cORF的长度以及氨基酸序列:
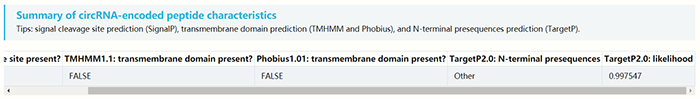
提供的内容包括cORF的起始和终止位置,cORF的长度以及氨基酸序列: 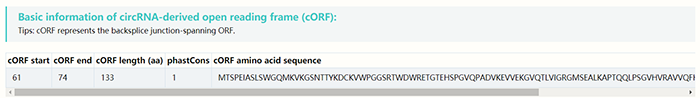 针对所编码多肽的总结,包括是否存在剪切,是否在胞膜上存在结构域等:
针对所编码多肽的总结,包括是否存在剪切,是否在胞膜上存在结构域等: 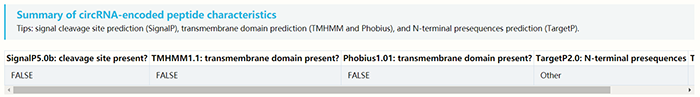
 所编码多肽的亚细胞定位:
所编码多肽的亚细胞定位: 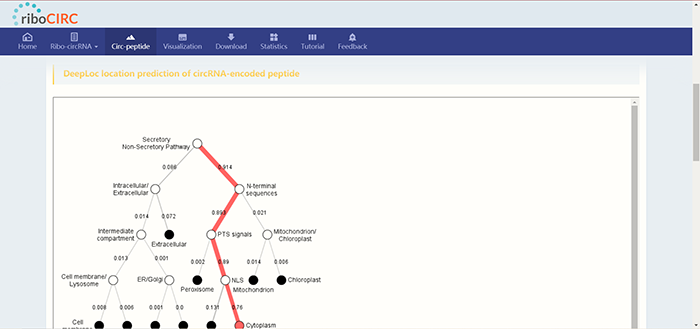 以及所编码多肽的结构预测。其中点击“result.tar.bz2”可以直接下载预测结果。 Conf.Score越高说明预测的二级结构越可靠。
以及所编码多肽的结构预测。其中点击“result.tar.bz2”可以直接下载预测结果。 Conf.Score越高说明预测的二级结构越可靠。 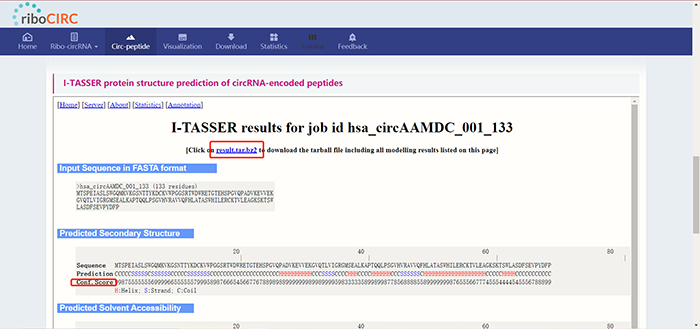 B值为负数说明残基在结构中更稳定:
B值为负数说明残基在结构中更稳定: 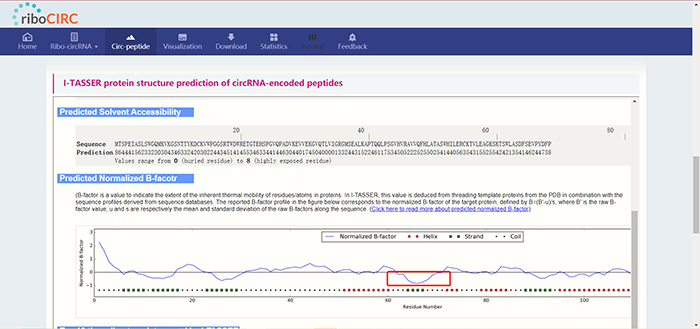 页面最后还提供了三级结构的预测模型,其中C值大于-1.5提示模型预测质量很好:
页面最后还提供了三级结构的预测模型,其中C值大于-1.5提示模型预测质量很好: 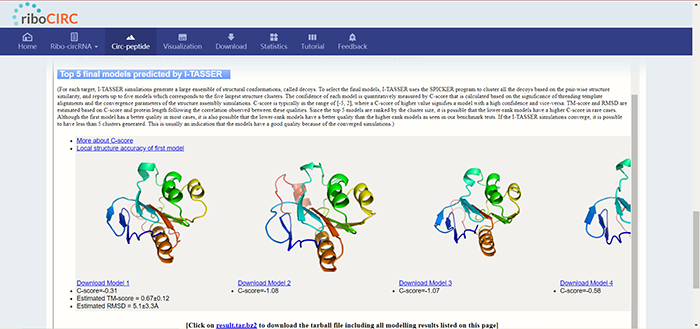 好啦,riboCIRC数据库的介绍我们就先进行到这里啦~这个数据库的优点显而易见,首先就是它足够的新,3月份才上线,收录了最新的数据,毕竟人家自己也做出了承诺会经常更新哈哈:
好啦,riboCIRC数据库的介绍我们就先进行到这里啦~这个数据库的优点显而易见,首先就是它足够的新,3月份才上线,收录了最新的数据,毕竟人家自己也做出了承诺会经常更新哈哈:  其次就是相较于Transcirc数据库,虽然它收录的编码证据没有这么多,但是在别的方面,包括可视化,引物设计,多肽结构分析等方面更胜一筹。目前使用该数据库的人还很少,还不赶紧吃一波红利?
其次就是相较于Transcirc数据库,虽然它收录的编码证据没有这么多,但是在别的方面,包括可视化,引物设计,多肽结构分析等方面更胜一筹。目前使用该数据库的人还很少,还不赶紧吃一波红利?






附件